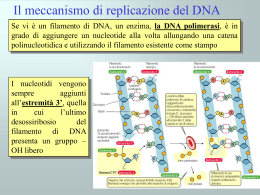
DUPLICAZIONE del DNA Modello di Watson-Crick 1. molecola composta da due catene di nucleotidi 2. le due catene si avvolgono a spirale intorno ad un asse centrale formando una doppia elica destrorsa (senso orario discendente) 3. lo scheletro Zucchero-fosfato-zucchero e’ situato all’esterno della molecola con le basi rivolte verso l’esterno 4. Le basi sono quasi perpendicolari all’asse della molecola, impilate una sull’altra 5. le due catene sono tenute insieme da legami idrogeno Modello di Watson-Crick 6. La pirimidina di una catena è sempre accoppiata alla purina di un’altra catena (C=G ; A=T) 7. Le due catene formate dalla doppia elica corrono in direzioni opposte (antiparallele) 8. L’avvolgimento del DNA genera 2 solchi di grandezza differente (solco maggiore e solco minore). 9. La doppia elica forma un giro completo ogni 10 basi (3,4 nm) 10.Le due catene sono fra loro complementari Modello di Watson-Crick Importanza del modello di Watson-Crick 1. DEPOSITO DELL’INFORMAZIONE GENETICA 2. AUTOREPLICAZIONE ED EREDITARIETA’ 3. ESPRESSIONE DEL MESSAGGIO GENETICO °°°°°°°°°°°°°°°°°° 1. e 2. si basano sulla stabilità del DNA e sulla capacità di una catena di servire da stampo per la costruzione di una nuova catena di DNA 3. Si basa sull’organizzazione del DNA in GENI in regioni di controllo; trascritte/tradotte; terminazione DUPLICAZIONE del DNA La duplicazione del DNA avviene nella fase S del ciclo cellulare MITOSI 4n 2n 1n G1………S………G2………M………G1………S………G2………M……… M G1 G2 G0 S DUPLICAZIONE del DNA La duplicazione del DNA avviene nella fase S del ciclo cellulare MEIOSI 4n e FECONDAZIONE 2n 1n G1………S………G2………M1..…M2………XX………S………M………S.. M1/M2 FECONDAZIONE G2 G1 G0 S REPLICAZIONE SEMICONSERVATIVA I due filamenti parentali si separano e ciascuno serve da stampo per la sintesi di un nuovo filamento figlio mediante accoppiamento di basi no! no! ORIGINE DI REPLICAZIONE porzione di circa 250 bp con sito di attacco per le proteine necessarie ad iniziare la replicazione e consentono l’accesso all’elicasi Si attivano una sola volta ad ogni ciclo cellulare (fase S) Si attivano in successione in regioni variabili del genoma Le elicasi sono enzimi che catalizzano lo svolgimento del DNA parentale Le topoisomerasi sono enzimi che catalizzano la rottura e la riunione reversibile dei filamenti di DNA parentale Tipo I taglia un solo filamento Tipo II taglia entrambi i filamenti DNA POLIMERASI 1.Sintetizzano il DNA solo in direzione 5’--> 3’ aggiungendo dNTP al gruppo idrossile in 3’ di una catena in crescita 2.Funzionano solo su uno stampo di DNA 3.Aggiungono un nuovo dNTP soltanto ad un filamento “PRIMER” preformato che forma legami idrogeno con lo stampo DNA POLIMERASI Le DNA polimerasi non possono iniziare la sintesi di DNA partendo da dNTP liberi “PRIMER” Il primer viene sintetizzato da una RNA polimerasi che non necessita di primers DNA POLIMERASI BATTERI : TIPO I TIPO II TIPO III --------------------------------------EUCARIOTI : α β γ --> Mitocondriale δ ε DNA POLIMERASI DNA polimerasi α, δ, ε attive in cellule in divisione/replicazione °°°°°°°°°°°°°° DNA polimerasi β sempre attiva sia in cellule quiescenti che in divisione ha un ruolo nella riparazione dei danni al DNA DNA POLIMERASI ATTIVITA’ di “PROOFREADING” delle DNA polimerasi durante l’allungamento la DNA polimerasi si destabilizza con una mutazione TAGCGTG ……ATCGCGCTGATAGATGCTGGTGATGATGGGGATTTTATATATAATATA TAAGGA… La DNA polimerasi torna indietro (3’-->5’) con ATTIVITA’ ESONUCLEASICA rimuove le basi errate sostituendole con le basi corrette (perdita del 3xfosfato in 5’) DNA POLIMERASI FEDELTA’ della REPLICAZIONE - le DNA polimerasi possono riparare errori o danni al DNA. Durante la sintesi le DNA pol α, ε (pol III nei batteri) vedono le mutazioni e le riparano T TAGCG GACTATC ……ATCGCGCTGATAGATGCTGGTGATGATGGGGATTTTATATATAATATA TAAGGA… Il loop viene riconosciuto e rimosso dalla DNA pol ε DNA doppia catena antiparallela la DNA polimerasi non puo’ sintetizzare il secondo filamento che corre in direzione opposta perche’ non puo’ andare in 3’-->5’ X Generazione del: FILAMENTO LEADER sintetizzato in modo continuo dalla DNA pol in direzione 5’-->3’ FILAMENTO RITARDATO sintetizzato tramite i FRAMMENTI DI OKASAKI in modo discontinuo e successivamente uniti tra loro La Primasi (batteri) o la DNA pol α/primasi (eucarioti), enzima che sintetizza brevi frammenti di RNA complementari allo stampo del filamento ritardato sulla forcella di replicazione Fornisce il “primer” di innesco per la DNA pol δ che forma il frammento di Okasaki il primer deve essere rimosso e sostituito La rimozione e sostituzione del primer e’ operata: - nei batteri --> dalla RNAsi H - negli eucarioti --> dalla azione RNAsica delle DNA polimerasi Attivita’ 5’-3’ ESONUCLEASICA Il primer viene eliminato e la parte di RNA rimossa viene riempita dall’azione riparatrice della DNA pol δ (DNA pol III nei batteri) Al termine i frammenti di Okasaki contigui formatesi sul filamento ritardato Vengono legati utilizzando ATP dalla DNA LIGASI TELOMERASI Aggiungono i “telomeri” alle estremita’ dei cromosomi Agiscono senza stampo di DNA La Telomerasi e’ una Trascrittasi inversa Sintetizza DNA da uno stampo di RNA contenuto nell’enzima (coenzima) Filmati sulla replicazione del DNA(ed altro) alla pagina: http://www.wehi.edu.au/wehi-tv/dna/
Scarica