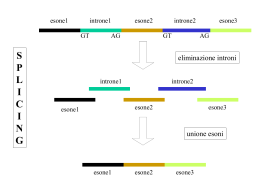
CORSO DI LAUREA SPECIALISTICA IN BIOTECNOLOGIE DEL FARMACO Adriana Maggi BIOTECNOLOGIE FARMACOLOGICHE LEZIONE 3 ESPRESSIONE GENICA IN EUCARIOTE: SEQUENZA DI EVENTI 3’ RNA, trascritto primario TRASCRIZIONE MODIFICAZIONI TP Capping e poliadenilazione Splicing o maturazione Fuoriuscita dal nucleo riconoscimento da parte dei ribosomi traduzione Modificazioni post-traduzionali TRADUZIONE ESPRESSIONE GENICA Trascrizione (trascrittoma) Il trascrittoma è mantenuto dalla cellula madre alla cellula figlia ed è modificato a seconda degli stimoli esterni Traduzione (proteoma) Contenuto di RNA in cellula eucariota 20-30 pg – 1% dell’intera cellula 0,05-01 pg in cellule batteriche) TOTAL RNA CODING 4% transcriptome PRE-mRNA mRNA NON CODING 96% Pre-rRNA Pre-tRNA snRNA snoRNA scRNA rRNA tRNA PRE-mRNA END MODIFICATIONS mRNA FUNCTIONS - CAP Transport to cytoplasm, initiation of translation - polyA Termination transcription, init transl, turnover Splicing No role other than removal of introns Alternative splicing Enables one RNA to code for two proteins RNA editing Enables one RNA to code for two proteins Converts abbreviated transcripts in functional RNAs Places termination codons in some mitoch mRNAs 5'-Capping Capping occurs shortly after transcription begins. m7G is linked to the first nucleotide by a special 5'-5' triphosphate linkage. In most organisms, the first nucleotide is methylated at the 2'-hydroxyl of the ribose. In vertebrates, the second nucleotide is also methylated. 3'-Polyadenylation A stretch of adenylate residues are added to the 3' end. The poly-A tail contains ~ 250 A residues in mammals, and ~ 100 in yeasts Polyadenylation at the 3' end. The major signal for the 3' cleavage is the sequence AAUAAA. Cleavage occurs at 10-35 nucleotides downstream from the specific sequence. A second signal is located about 50 nucleotides downstream from the cleavage site. This signal is a GU-rich or U-rich region. MATURAZIONE DEL TRASCRITTO PRIMARIO O EXCISIONE (SPLICING) DEGLI INTRONI A FORMARE RNAm ESISTONO QUATTRO CLASSI DI INTRONI : • INTRONI Self-Splicing DI GRUPPO I • INTRONI Self-Splicing DI GRUPPO II • INTRONI DI RNA NUCLEARE BERSAGLIO DELL’APPARATO DI SPLICING (Spliceosomal Introns) • INTRONI tRNA Nucleare che subiscono matutrazione per via enzymatica Tom Cech Sidney Altman Tom Cech e Sidney Altman hanno ricevuto il premio Nobel per la Chimica ne 1989 per la scoperta del meccanismo di self splicing in Tetrahymena e per la scoperta che la componente catalitica della ribonucleasi P responsabile per la maturazioen del tRNA in E.coli era una molecola di RNA. INTRONI Self-Splicing DI GRUPPO I 1. Nucleophilic attack by the 3' OH group of a free guanine nucleoside on the 5' phosphorus atom at the 5' end of the intron. 2. The 3'OH group at the 3' end of the upstream exon carries out a nucleophilic attack on the phosphorus atom at the 5' end of the downstream exon. INTRONI Self-Splicing DI GRUPPO I In questa classe di introni che comprende l’introne di rRNA di Tetrahymena lo splicing dipende dalla struttura terziaria della molecola. Perché avvenga il “self-splicing” Even though we often think of them as singlela molecola di RNA deve avere una struttura secondaria e terziaria ben definita; alcune variazione sono consentite ma solo se i cambiamenti mantengono i siti di splicing 3’ e 5’ molto prossimi l’uno all’altro e la guanosina catalitica nelle immediate vicinanze. Introni I tipo Introni I tipo MECCANISMO DI EXCISIONE INTRONI DI TIPO II 1° reazione di transesterificazione promossa da C 2’ adenosina Rottura legame fosfodiestereo Formazione nuovo legame 5’-2’ che lega primo nt con adenosina interna Formazione del lariate e riformazione del legame fosfodiestereo Sequences, secondary structure and tertiary interactions of group II intron bI1 from yeast mitochondria Black boxes, exon sequences; continuos line, six major intron domains (D1-D6); 5’GUGAG...AU3’, consensus nucleotides at the intron boundaries. Red line, ID3 subdomain; dashed red lines, interactions of ID3 with intron and exon sequences, (EBS1/IBS1 and a-a´). Marked in blue, individual nucleotides and structures involved in tertiary interactions (EBS2-IBS2, e-e´, dd´, g-g´ and z-z´). Note, for bI1 the d-d’ interaction (U-U) is not established. Introni self-splicing di gruppo II I prodotti di razione sono la molecola di RNA correttamente maturata e un introne che forma una struttura detta lariate. Come per gli introni di gruppo I anche per questa classe di introni una corretta struttura secondaria e terziaria è indispensabile per lo splicing. IN CONCLUSIONE LE DIFFERENZE TRA INTRONI DI GRUPPO I E II SONO: • L’AGENTE RESPONSABILE DELL’ATTACCO NUCLEOFILO INIZIALE. Spliceosomal Intron Splicing I passaggi di base necessari per la maturazione di RNAtrascritto primario in eucariote sono gli stessi visti per il gruppo II di introni self-slicing e i prodotti di reazione anche in questo caso sono l’RNA maturato correttamente e un lariate. A differenza degli introni self-splicing la reazione in eucariote richiede la partecipazione di un sistema elaborato di altri fattori quali le small nuclear ribonucleoprotein particles or snRNPs. Ogni snRNP è un complesso di diverse proteine e piccoli U-RNA detti small nuclear RNA or snRNA. Gli snRNA coinvolti nello splicing sono cinque INTRONI DI RNA NUCLEARE BERSAGLIO DELL’APPARATO DI SPLICING 5’ Ruolo snRNP e proteine associate nello splicing 1 3’ 5’ 3’ 5’ 3’ Commitment complex (E complex) U1 lega il sito di taglio in 5’ e U2AF lega il sito polipirimidinico Complex A U2 lega il sito “branch” Complex B si associano U5, U6, U4 Ruolo snRNP e proteine associate nello splicing 2. Riposizionamento, U1 si dissocia, U5 si associa all’introne; U 6 lega il sito di taglio in 5’ Complex C U4 si dissocia, U6 e U2 catalizzano la transesterificazione U5 l’esone al sito di taglio in 3’ Avviene il taglio in 5’ e si forma il lariate Avviene il taglio in 3’ e la ligazione Tra gli esoni LE SEQUENZE DI SPLICING SONO CONSERVATE NEI VERTEBRATI 5’ splice site: 5’-GU-3’ Tratto polipirimidinico 3’ splice site 5’-AG-3’ Esistono introni AU-AC sono simili a introni GU-AG ma richiedono Un apparato di splicing differente RNA EDITING Modificazioni enzimatiche tramite cui nucleotidi di RNAm vengono modificati RNA EDITING •Editing by deamination •Editing by insertion or deletion RNA EDITING RNA editing: specific nucleotides are modified to change one nucleotide into another. One example is the de-amination of cytidine which occurs in mammalian apolipo protein B mRNA in the intestine. Here, a specific C is changed into a U, introducing a stop codon for translation and thereby a shorter version of the protein. A second example is de-amination of adenosine to inosine. Inosine is read as guanosine by the translation machinery. The adenosine is present in a double stranded region of the mRNA and the enzyme adenosine de-aminase that acts on RNA, ADAR, catalyzes the reaction. Examples of mRNAs edited by ADAR are mRNA encoding glutamate receptors, serotonin receptors and potassium channels in the central nervous system and hepatitis delta virus RNA. GUIDE RNA The mitochondria for some trypanosome protozoa undergo gRNA-directed mRNA editing. The gRNA identifies particular sequences and directs the insertion or deletion of uridylates (U) from the mRNA by enzymes residing in a macromolecular complex. The edited portion of the mRNA is in the coding region, which has the effect of modifying the protein that is produced. ESPRESSIONE GENICA IN EUCARIOTE: SEQUENZA DI EVENTI 3’ RNA, trascritto primario TRASCRIZIONE MODIFICAZIONI TP Capping e poliadenilazione Splicing o maturazione Fuoriuscita dal nucleo riconoscimento da parte dei ribosomi traduzione Modificazioni post-traduzionali TRADUZIONE TRADUZIONE DI mRNA IN PROTEINE CELLULE EUCARIOTE MODIFICAZIONI POST-TRADUZIONALI DI PROTEINE Proteolisi Glicosilazione Acilazione metilazione Fosforilazione Sulfunilazione Prenilazione
Scaricare