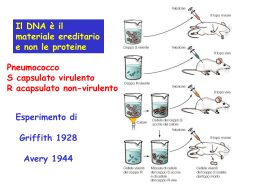
Enzimi di restrizione • riconoscono sequenze specifiche, in genere di 4-6 nucleotidi, di DNA a doppio filamento. • Queste sequenze sono generalmente palindromi, cioè identiche se lette in direzione 5'--> 3' sia su un filamento che sull'altro. -Servono a tagliare il DNA in modo preciso e prevedibile -Sono stati isolati più di 1200 enzimi da procarioti -Riconoscono più di 130 diverse sequenze nucleotidiche (sequenze palindromiche di 4, 6 o 8 nucleotidi). Il nome deriva dal batterio da cui è stato isolato: EcoRI E = genere Escherichia co = specie coli R = ceppo RY13 I = prima endonucleasi isolata BamHI B = genere Bacillus am = specie amyloliquefaciens H = ceppo H I = prima endonucleasi isolata GAATTC CTTAAG GGATCC CCTAGG Simmetria binaria Tipi di taglio • • • • -BLUNT ENDS O TAGLIO NETTO Es. SmaI 5’-CCC GGG-3’ 5’-CCC-3’ 5’- GGG-3’ 3’-GGG CCC-5’ 3’-GGG-5’ 3’-CCC-5’ • -STICKY ENDS O CODINE ADESIVE: • -5’ PROTRUDING (5’ SPORGENTE) • -3’ PROTRUDING (3’ SPORGENTE) – Es. Eco RI (5’ PROTRUDING) • 5’-G/AATTC-3’ • 3’-CTTAA/G-5’ 5’-G-3’ 5’-AATTC-3’ 3’-CTTAA-5’ 3’-G-5’ – Es. Kpn I (3’ PROTRUDING) • 5’-GGTAC/C-3’ • 3’-C/CATGG-5’ 5’-GGTAC -3’ 5’-C-3’ 3’-C-5’ 3’-CATGG-5’ Quante volte taglia un enzima • • • • • • Probabilità di trovare un sito di restrizione: basi probabilità 4 (1/4)4 = 1/256 5 (1/4)5 = 1/1024 6 (1/4)6 = 1/4096 8 (1/4)8 = 1/65536 DNA ligasi defosforilazione Gel elettroforesi Southern blot • Il Southern Blot è una tecnica che permette di identificare la presenza del frammento di DNA che a noi interessa tra una miscela di tantissimi frammenti. Chimica del DNA • Soluzione viscosa a pH 7 • Diminuzione viscosità a pH estremi o a T> 80°C • Denaturazione ( aumento D.O) • Rinaturazione (diminuzione D.O.) • Formazione di ibridi anche tra catene di specie diverse • I nucleotidi vanno incontro a trasformazioni chimiche (mutazioni) DENATURAZIONE E RINATURAZIONE La denaturazione del DNA consiste nella separazione completa dei due filamenti complementari che lo costituiscono. Il processo inverso è detto rinaturazione. ANALISI FISICA Maggiore è G+C, maggiore è Tm. 1.2 50 1.1 1.0 40 60 Tm 80 Temperatura (°C) 0 100 Grado di denaturazione (%) Temperatura di melting (Tm): Temperatura in corrispondenza della quale il DNA è denaturato al 50%. 1.3 Assorbanza a 260 nm Effetto ipercromico: Aumento dell’A260nm durante il processo di denaturazione del DNA. Curva di fusione CINETICA di RINATURAZIONE Curva C0t Effetto ipocromico: 0 Diminuzione dell’A260nm durante il processo di rinaturazione del DNA. 1) C0 2) Tempo Concentrazione espressa in nucleotidi/unità di volume Rinaturazione (%) Parametri che influenzano la rinaturazione: DNA a rinaturazione rapida 50 DNA a rinaturazione lenta 100 La misura della velocità di rinaturazione può fornire utili informazioni circa la complessità del DNA -2 -1 0 1 2 Log Cot 3 4 5 forma A forma B forma Z Senso elica destrorsa destrorsa sinistrorsa Diametro 26 A 20 A 12 A Coppie di basi per ogni giro di elica 11 10.5 12 Distanza tra basi 2.6 A 3.4 A 3.7 A Piegamento basi rispetto all’asse dell’elica 20° 6° 7° Conformazione dello zucchero C-2’ endo C-2’ endo per pirimidine C-3’ endo per purine anti anti per pirimidine sin per purine C-3’ endo Conformazione Legame glucosidico anti % ss DNA rimanente Cot Fig. 13 Curva di Cot SONDE E MARCATURA • La sonda è un frammento specifico di DNA (o RNA), reso radioattivo, capace di riconoscere una sequenza complementare di DNA o RNA in una popolazione di tanti frammenti. Metodi di marcatura • Nick transaltion • Random priming • Marcatura terminale nick con un -OH disponibile 5’P 3’OH L’attività 3’5’ esonucleasica rimuove il nucleotide 5’P 3’OH L’attività 5’3’ polimerasica sostituisce il nucleotide 5’P 3’OH L’attività 5’3’ esonucleasica sposta il nick verso il 3’ 5’P 3’OH -3’OH -5P’ -3’OH -5P’ -3’OH -5P’ -3’OH -5P’ 5’P -3’OH 3’OH -5P’ Denaturazione ed appaiamento degli esanucleotidi casuali 5’P -3’OH 3’P 5’OH Enzima Klenow + dNTP* 5’P -3’OH 3’P 5’OH MARCATURA delle SONDE OLIGONUCLEOTIDICHE RANDOM PRIMING Metodo dell’innesco casuale 5’3’- NICK TRANSLATION Spostamento dell’incisione -3’ DNA -5’ denaturazione 3’-TAGCAG-5’ aggiunta di inneschi esanucleotidici 3’-GCATGC-5’ 5’-TGCAGT-3’ -3’ 5’3’-TAGCAG-5’ -5’ Frammento di Klenow, dNTP 3’-TAGCAG-5’ 5’-GCATAC-3’ 3’- DNA sonda marcato 5’3’- GCAAG CGCGTTC GC denaturazione 5’3’- -5’ -3’ -5’ riempimento del buco prodotto con un nucleotide radioattivo e rimozione del nucleotide successivo CAAG CGCGTTC GC G -3’ -5’ il taglio si muove in direzione 5’-3’ AAG CGCGTTC -3’ -5’ 32P-dCTP -5’ G -3’ DNA -5’ taglio di un’elica e rimozione di un nucleotide mediante la DNA polimerasi I dGTP -3’ 5’-TGCAGT-3’ 5’3’- Sintesi DNA 5’3’-TAGCAG-5’ CGCGTTC 32P-dCTP 5’-GCATAC-3’ dATP 32P-dCTP dTTP dGTP 3’- 5’3’- 3’-TAGCAG-5’ 3’- GCGCAAG ibridizzazazione 5’-GCATAC-3’ 5’-TGCAGT-3’ 5’3’- GC G C AG CGCGTTC -3’ -5’ 5’P -3’OH 3’OH -5P’ Trattamento con fosfatasi 5’OH -3’OH 3’OH -5’OH Polinucleotide chinasi+ [32P]dATP 5’P 3’OH -3’OH -5P’ • I tipi di marcatura non radioattiva sono essenzialmente: • -marcatura isotopica diretta (incorporazione di nucleotidi modificati conteneti un fluorocromo, cioè un gruppo chimico in grado di emettere fluorescenza se esposto alla luce di una certa l); • -marcatura indiretta ( associazione tra una molecola indicatrice e un precursore nucleotidico). • I sistemi più usati sono: • -Il sistema biotina-streptavidina -La digossigenina O Uracile O NH HN PRINCIPIO DELLE SONDE NON RADIOATTIVE: Affinità molto elevate tra Biotina e Avidina o Streptavidina Uracile HN S dUTP O N Biotina Desossiribosio Trifosfato B B B B C U C G A G A U G U C A U G A G C T C T A C A G T A DNA sonda DNA target Ibridazione del DNA sonda col DNA bersaglio dei cromosomi (A) Avidina-enzima E A E A B B A U G U C T A C A G (B) Avidina-fluoroforo E = molecole reporter fosfatasi alcalina o b-galattossidasi F = molecole reporter F A F A B B A U G U C T A C A G Ibridazione • L’ibridazione è quel processo per cui due filamenti di DNA (o RNA) possono appaiarsi e formare un ibrido (doppia elica) più o meno stabile mediante la formazione di legame idrogeno per un certo numero di basi complementari. • L’ibrido può formarsi tra DNA/DNA e DNA/RNA. • L’ibridazione può avvenire anche tra molecole di DNA non perfettamente complementari. • Sono molto importanti le condizioni sperimentali. Condizioni stringenti • Vengono definite condizioni stringenti, quelle condizioni sperimentali che permettono la migliore ibridazione. • Condizioni di ibridazione piu’ stringenti (richiedono maggiore percentuale di omologia tra sonda e bersaglio): – maggiore Temperatura – minore concentrazione salina – presenza di denaturanti chimici • Condizioni di ibridazione meno stringenti (permettono minore percentuale di omologia tra sonda e bersaglio): – minore Temperatura – maggiore concentrazione salina – assenza di denaturanti chimici Mappe di restrizione in diagnostica ...MAPPATURA di RESTRIZIONE TRATTAMENTO Nessuna digestione Enzima A DIMENSIONE dei FRAMMENTI (Kb) INTERPRETAZIONE 9 9 A 2+7 2 7 A B Enzima B 3 3+6 6 B A 3 6 A B 1+2+6 2 Enzima A+ B 1 B A 2+3+4 2 4 6 3 Sequenziamento con isotopi radioattivi a = adenina c = citosina g = guanina t = timina g t a c gtacg tacg tac MICROSATELLITE ripetizione: adenina e citosina acacacacacacacacacacacacacacac dinucleotide :(ac)15 1) ESTRAZIONE DEL DNA Sangue Peli Seme DNA Doppia Elica 2) PCR Polymerase Chain Reaction Individuo A 3 paia di cromosom i omologhi 3) ELETTROFORESI Individuo B Individuo C campione di controllo Sequenziatore Automatico Elettroferogramma di un campione con 12 microsatelliti Immagine Computerizzata dell’ elettroforesi PADRE MADRE GENOTIPO: 263/263 GENOTIPO: 263/277 DIAGNOSI POSITIVA FIGLIO GENOTIPO: 263/263 FIGLIO GENOTIPO: 263/277 Profilo genetico di un campione PADRE GENOTIPO: 263/263 GENOTIPO: 128/140 PADRE 128 263 GENOTIPO: 128/146 GENOTIPO: 263/277 MADRE MADRE 128 263 146 277 FIGLIO 263 GENOTIPO: 128/128 128 GENOTIPO: 263/263 FIGLIO 140 FIGLIO GENOTIPO: 124/128 124 128 GENOTIPO: 263/277 FIGLIO 263 277 FIGLIO GENOTIPO: 128/128 128 DIAGNOSI POSITIVA DIAGNOSI NEGATIVA
Scarica