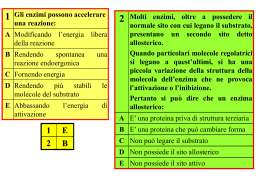
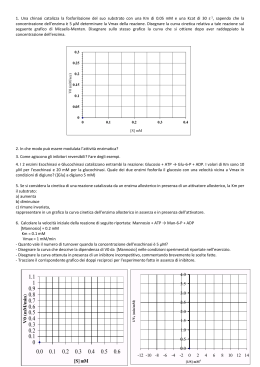
ENZIMI CATALISI ENZIMATICA. Meccanismo d’azione degli enzimi; Energia dl attivazione; Componenti strutturali e funzionali; Nomenclatura e classificazione; Specificità dl substrato ed azione; Modello Lock and Key; Coenzimi principali; Principio di additività dell’energia libera in reazioni accoppiate. ενζυµον (“nel lievito”): un po’ di storia... • Nell’antichità, denominati fermenti – Diastasi del malto idrolizza l’amido più velocemente dell’acido solforico • • • • • • • • • • • 1835: Prima teoria della catalisi chimica (Berzelius) 1860: Fermentazione causata da sostanze termolabili (Pasteur) 1877: Primo uso del termine “enzima” 1897: Estrazione dal lievito degli enzimi della fermentazione alcolica, dimostrazione che può avvenire in assenza di cellule integre (Buchner) 1913: Equazione di Michaelis-Menten, primo modello matematico del funzionamento degli enzimi 1926: Cristallizzazione di ureasi (Sumner) 1930: Cristallizzazione di pepsina, tripsina e chimotripsina: tutti gli enzimi sono proteine (Northrop) 1965: Teoria dell’allosterismo (Jacob, Monod, Changeaux) 1992: L’attività degli enzimi può essere regolata con modificazioni covalenti (Fischer, Krebs) 1997: Anche RNA può agire come catalizzatore: non tutti gli enzimi sono proteine 2004: Ruolo dell’ubiquitina nella distruzione degli enzimi (Ciechanover, Hershko, Rose) 1 Alcune caratteristiche della materia vivente • Composta da molecole non viventi, vale più della somma delle sue parti (1+1=3) • Si presenta con poche differenze in specie diverse • Ogni componente con funzione specifica e insostituibile, più funzioni per ciascun componente • Trasforma l’energia derivata dall’ambiente costruendo strutture ordinate e complesse da materiale disordinato e semplice • Abilità di autoriprodursi in modo preciso utilizzando le informazioni contenute nel proprio interno • Capacità evolutiva, irritabilità ed adattamento all’ambiente • Resa ottimale: – Svolge solo le reazioni necessarie minimizzando la produzione di prodotti di scarto – Impedisce reazioni indesiderate prevenendo il dispendio energetico – Porta a termine le reazioni in tempi brevi – E’ indipendente dalle condizioni ambientali (temperatura, pressione) – Può riutilizzare la stessa “macchina” per molte volte Energia di attivazione • Y si trova ad uno stato energetico inferiore rispetto a X – Conversione X→Y termodinamicamente favorevole • Ma la reazione non può avvenire se X non acquisisce sufficiente energia per superare la barriera dell’energia di attivazione 2 Numero di molecole Distribuzione delle molecole a vari livelli di energia Energia di attivazione Molecole con energia maggiore dell’energia di attivazione Energia Aumento di temperatura Presenza di un catalizzatore Energia Energia di attivazione A Energia di attivazione senza catalizzatore Stato iniziale Riduzione dell’energia di attivazione del substrato mediante: Combinazione transitoria col substrato Stabilizzazione dello stato di transizione Inserimento di numerosi stati di transizione con livello energetico inferiore Energia di attivazione con catalizzatore Energia liberata dalla reazione P Stato finale 3 Un enzima immaginario: la metallasi • La barretta deve essere piegata prima di essere rotta (stato di transizione) • Enzima con alta affinità per il substrato: stabilizza il substrato • Enzima con alta affinità per lo stato di transizione: stabilizza lo stato di transizione e accelera la reaziuione Enzimi: catalizzatori biologici • Non modificano l’equilibrio della reazione, ma solo la velocità con la quale l’equilibrio viene raggiunto • Non vengono modificati nella reazione, e al termine della reazione sono subito disponibili per catalizzare una nuova reazione 4 H2O2 e catalasi 2 H2O2 ⇐⇒ 2 H2O + O2 Catalizzata da catalasi Reazione spostata a destra (Keq alta) ma molto lenta (k velocità bassa) DNA RNA ribosomi Struttura generale degli enzimi Zimogeno (precursore) Substrato Apoenzima Prodotto Sito attivo ENZIMA ATTIVO Ubiquitina Lisosomi degradazione Parte non-proteica ione metallico o coenzima Sito allosterico Effettore allosterico 5 Nomenclatura degli enzimi • Denominazione classica costituita da 3 parti: – Nome del substrato – Nome del coenzima – Nome della reazione catalizzata + “asi” Esempio: Lattico-NAD-deidrogenasi • International Enzyme Commission, fondata nel 1956 da Prof. M. Florkin: – 77 membri, presidente attuale Angelo Azzi – Classificazione degli enzimi secondo il sistema delle classi EC Esempi: Creatin kinasi: EC 2.7.3.2, adenosintrifosfato : creatin N-fosfo trasferasi Lattato deidrogenasi: EC 1.1.1.27, lattato : NAD+ ossidoreduttasi – Ha funzionato bene fino agli anni ‘90 (circa 3200 enzimi) • Progetto genoma umano (HUGO): – Esistono circa 20,000 geni, in buona parte enzimi Classificazione internazionale degli enzimi • 1 - ossidoreduttasi: reazioni di ossido-riduzione – – – – – – – – – 1.1 agisce sul gruppo CHOH del donatore 1.2 agisce sul gruppo aldeidico o chetonico del donatore 1.3 agisce sul gruppo CH-CH del donatore 1.4 agisce sul gruppo CH-NH2 del donatore 1.5 agisce sul gruppo C-NH del donatore 1.6 agisce su NADH2 o NADPH2 1.7 agisce su altri composti azotati 1.8 agisce su gruppi solforati 1.9 agisce sui gruppi eme 6 Classificazione internazionale degli enzimi • 1 - ossidoreduttasi: reazioni di ossido-riduzione • 2 - transferasi: trasferimento di gruppi chimici da una molecola ad un'altra – – – – – – – – 2.1 gruppi ad una unità di carbonio 2.2 gruppi chetonici o aldeidici 2.3 gruppi acilici 2.4 gruppi glicosidici 2.5 gruppi alchilici 2.6 gruppi azotati 2.7 gruppi fosforici 2.8 gruppi contenenti zolfo Classificazione internazionale degli enzimi • 1 - ossidoreduttasi: reazioni di ossido-riduzione • 2 - transferasi: trasferimento di gruppi chimici da una molecola ad un'altra • 3 - idrolasi: reazioni di idrolisi – – – – – – – 3.1 legami esterei 3.2 legami glicosidici 3.3 altri legami 3.4 legami peptidici 3.5 legami C-N non peptidici 3.6 legami anidridici 3.7 legami C-C 7 Classificazione internazionale degli enzimi • 1 - ossidoreduttasi: reazioni di ossido-riduzione • 2 - transferasi: trasferimento di gruppi chimici da una molecola ad un'altra • 3 - idrolasi: reazioni di idrolisi • 4 - liasi: reazioni di addizione a doppi legami – – – – 4.1 legami C = C 4.2 legami C = O 4.3 legami C = N 4.4 legami C = S Classificazione internazionale degli enzimi • 1 - ossidoreduttasi: reazioni di ossido-riduzione • 2 - transferasi: trasferimento di gruppi chimici da una molecola ad un'altra • 3 - idrolasi: reazioni di idrolisi • 4 - liasi: reazioni di addizione a doppi legami • 5 - isomerasi: reazioni di trasformazione di una molecola nel suo isomero – 5.1 racemasi – 5.2 cis-trans isomerasi 8 Classificazione internazionale degli enzimi • 1 - ossidoreduttasi: reazioni di ossido-riduzione • 2 - transferasi: trasferimento di gruppi chimici da una molecola ad un'altra • 3 - idrolasi: reazioni di idrolisi • 4 - liasi: reazioni di addizione a doppi legami • 5 - isomerasi: reazioni di trasformazione di una molecola nel suo isomero • 6 - ligasi: reazioni di formazione di nuovi legami con rottura di ATP in AMP e pirofosfato o ADP e Pi – – – – 6.1 legami C-O 6.2 legami C-S 6.3 legami C-N 6.4 legami C-C Classificazione internazionale degli enzimi • 1 - ossidoreduttasi: reazioni di ossido-riduzione • 2 - transferasi: trasferimento di gruppi chimici da una molecola ad un'altra • 3 - idrolasi: reazioni di idrolisi • 4 - liasi: reazioni di addizione a doppi legami • 5 - isomerasi: reazioni di trasformazione di una molecola nel suo isomero • 6 - ligasi: reazioni di formazione di nuovi legami con rottura di ATP in AMP e pirofosfato o ADP e Pi 9 Modello Lock and Key: l’enzima si combina chimicamente col substrato • Emil Fisher 1894 • Il sito attivo è costituito dal “negativo” del substrato – Si formano legami “esatti” fra enzima e substrato • Riconoscimento anche tridimensionale – Esempio: glicerol kinasi (lega solo il glicerolo in forma alfa) – Può distinguere stereoisomeri D- e L- degli aminoacidi • Spiega la specificità, ma NON spiega la stabilizzazione dello stato intermedio Un modello più sofisticato: induced fit + substrate strain • Daniel Koshland 1958 (University of California Berkeley) • Sito di legame non completamente pre-formato • L’interazione fra substrato e enzima induce un cambiamento conformazionale nell’enzima che prelude ad un legame più stabile col substrato (induced fit) • Il substrato è forzato verso la formazione dei prodotti (substrate strain) 10 Il legame enzima-substrato avviene tramite l’interazione delle cariche del substrato con alcuni aminoacidi del sito attivo • Esempio: proteasi a serina • Alterazioni della struttura primaria di un enzima possono mutarne l’attività causando malattie genetiche Il legame enzima-substrato induce un cambiamento conformazionale nell’enzima Esempio: esochinasi senza e con glucosio 11 Il cambiamento conformazionale forza il substrato ad assumere una nuova struttura o conformazione Esempio: lisozima, che idrolizza i polisaccaridi Dalla sequenza degli aminoacidi alla struttura delle proteine • Struttura primaria (sequenza degli aminoacidi) – Sempre lineare • Struttura secondaria (conformazione della catena) – α-elica o β-a pieghe • Struttura terziaria (tridimensionalità della catena) – Relazioni locali o remote dei gruppi R – Proteine globulari o fibrose • Struttura quaternaria (interazioni fra più catene) 12 Struttura primaria e mutazioni genetiche • Tutte le funzioni delle proteine necessitano del riconoscimento fra zone o domini della proteina con altre molecole o parti di molecola: – Enzima-substrato, molecole di adesione-membrane, anticorpo-antigene… • Il dominio preposto al riconoscimento e la molecola da riconoscere devono essere complementari in termini di struttura e di carica • Il riconoscimento dipende dalla distribuzione dei gruppi R e dalla sequenza degli aminoacidi • Una mutazione della struttura primaria può impedire o rendere difficile il riconoscimento, causando disfunzioni più o meno gravi – Se ne deriva uno stato patologico, quella mutazione interessa il dominio di riconoscimento – Se la mutazione è silente (non genera malattie o disfunzioni), è possibile che l'aminoacido mutato non sia nel dominio di riconoscimento • Spesso è sufficiente la mutazione di un solo aminoacido • Tutte le malattie causate da tali mutazioni sono genetiche • Tutte le malattie genetiche originano una mutazione della struttura primaria delle proteine Di tutte le reazioni possibili per un substrato, l’enzima ne favorisce una sola • Gli enzimi forzano le vie metaboliche attraverso reazioni selezionate (ad es: A > B > H > K > N > T > Z) favorendo il flusso unidirezionale di materiale 13 Coenzimi • Piccole molecole organiche, spesso derivate da vitamine • Si legano all’enzima – Spesso fungono da “secondo substrato” • Determinano la specificità della reazione catalizzata Adenosin trifosfato (ATP) • Coenzima delle chinasi, trasferisce un gruppo Pi al substrato • Esempio: Glucosio + ATP ⇒ Glucosio-6-fosfato + ADP • In molte chinasi, il substrato vero è Mg-ATP 14 Nicotinamide Adenina Dinucleotide (NAD+) • Coenzima di deidrogenasi • Derivato da niacina • Trasferisce un protone da/a substrati: – lattato + NAD+ ⇐⇒ piruvato + NADH + H+ • Esiste una forma fosforilata NADP+ Flavina adenina dinucleotide (FAD) • Coenzima di deidrogenasi • Derivato da riboflavina (vit. B6) • Trasferisce due protoni da/a substrati – Succinato + FAD ⇐⇒ Fumarato + FADH2 15 Coenzima A • Trasportatore e attivatore di gruppi acilici (acidi grassi) e di acetato Metalli di transizione (Fe, Zn, Cu, Mn…) • Cofattori più che coenzimi • Presenti in 2/3 degli enzimi • Agiscono come stabilizzatori della proteina e come donatori/accettori di elettroni (acidi di Lewis), per esempio i citocromi 16 CINETICHE CHIMICHE E ENZIMATICHE Ordine di reazione; Ipotesi per l’azione degli enzimi; Equazione di Michaelis-Menten; Velocità massima e KM; Trasformazione di Lineweaver-Burk; Rappresentazione grafica delle reazioni enzimatiche. Cinetiche chimiche (A⇒P) 17 Effetto della concentrazione di A (A ⇒ P) A4 > A3 > A2 > A1 [prodotto] Velocità iniziale A4 A3 A2 A1 Tempo A1 A2 A3 A4 [A] Cinetiche chimiche vs cinetiche enzimatiche Velocità iniziale Cinetica enzimatica Cinetica chimica [A] 18 Ipotesi per l’azione degli enzimi [S] >> [E] E + S ⇐⇒ ES ⇒ EP ⇒ E + P • L’enzima si lega col substrato per formare ES: reazione reversibile • Il complesso ES subisce il cambiamento conformazionale che induce la formazione di P (reazione relativamente lenta) • L’enzima rilascia P e torna nello stato iniziale • La velocità globale è regolata da [ES] Ordine di reazione • Primo ordine A⇒P v=k[A]1, n=1 • Secondo ordine A+B⇒P v=k[A]1[B]1, n=2 • Pseudo primo ordine A+B⇒P v=k[A]1[B]1, n=2 ma se [B]»[A], v=k[A]1, n=1 • Ordine zero v=k, n=0 v dipende dalla concentrazione del catalizzatore 19 Interazione enzima-substrato v0 Se [S] è molto alto, v0 non cambia all’aumentare di [S] Vmax Ordine 0 Ordine misto 1° ordine Se [S] è alto, v0 è controllata da [E] Se [S] è intermedio, v0 è controllata sia da [S], sia da [E] Se [S] è basso, v0 è controllata da [S] [substrato] Commission on Enzyme Nomenclature of the International Union of Biochemistry • Attività enzimatica: moli di substrato convertito per unità di tempo in condizioni standard di temperatura, pH e forza ionica: – 1 katal = 1 mol/s – 1 IU = 1 micromole/min • Attività specifica: attività enzimatica per mg di proteina (moli/tempo/mg proteina) – Espressione di purezza • Costante catalitica o numero di turnover: attività enzimatica per mole di enzima (moli/tempo/mole enzima) 20 Equazione di Michaelis-Menten (1913) v0 = Vmax ⋅[S] K M + [S] Leonor Michaelis & Maud Menten Leonor Michaelis (Berlin, January 16, 1875 – New York, October 8, 1949). Maud Leonora Menten (Ontario, March 20, 1879 – July 26, 1960), among the first women in Canada to earn a medical doctorate. At that time women were not allowed to do research in Canada, therefore she decided to do research in other countries such as the United States and Germany. Accomplished musician and painter. Assunzioni di Michaelis-Menten • E + S ⇐⇒ ES ⇒ E + P • 1 solo substrato • Fattore limitante: formazione di ES • Tutto E ⇒ ES – Non esiste E libero – E saturo di S • ES stazionario 21 Ipotesi v0=Vmax/2 • KM = [S] quando v0=Vmax/2 • E’ una concentrazione (dimensioni moli/litro) • E’ indipendente da [E] v0 = Vmax ⋅[ S] K M + [S] Vmax Vmax ⋅ [S] = 2 K M + [S] 1 [S] = 2 K M + [S] K M + [S] = 2 ⋅[ S] K M = [S] Rappresentazione grafica delle reazioni enzimatiche v0 Vmax Vmax/2 KM [S] 22 Trasformazione di Lineweaver-Burk v0 = Vmax ⋅[S] K M + [S] Y 1 K M + [S] = v 0 Vmax ⋅ [S] 1 KM [S] = + v 0 Vmax ⋅ [S] Vmax ⋅ [S] 1 KM 1 1 = ⋅ + v 0 Vmax [S] Vmax X € Rappresentazione grafica delle reazioni enzimatiche 1 KM 1 1 = ⋅ + v0 Vmax [S] Vmax 1/v0 1/Vmax -1/KM 1/[S] 23 Rappresentazione grafica delle reazioni enzimatiche Grafico di Lineweaver-Burk V0 1/V0 Vmax Vmax/2 1/Vmax KM [S] -1/KM 1/[S] Esochinasi (HK) e glucochinasi (GK) Glucosio + ATP → Glucosio-6-fosfato + ADP • [glucosio] in sangue: 5 mM • [glucosio] intracellulare: 0.2-2 mM • Muscolo: HK – Inibita da Glucosio-6-fosfato – Utilizzo di glucosio indipendente da [glucosio] in sangue • Epatociti: GK – Non inibita da Glucosio-6-fosfato – Trasformazione di glucosio in glicogeno dipendente da [glucosio] in sangue • Pancreas: GK – Non inibita da Glucosio-6-fosfato – Sensore di [glucosio] in sangue per la produzione di insulina 24 Metabolismo dell’alcool Alcool deidrogenasi Aldeide deidrogenasi CH3-CH2OH ⎯⎯⎯⎯⎯→ CH3-CHO ⎯⎯⎯⎯⎯→ CH3-COO Etanolo Acetaldeide Acetato • KM di alcool deidrogenasi per etanolo: 1 mM (46 mg/l) – Enzima saturo dopo pochi drinks (anche uno) – Metabolizzazione segue una cinetica di ordine zero • • • – 3 varianti con poche differenze in termini di KM e Vmax KM di aldeide deidrogenasi per acetaldeide: 0.01 mM – Livello di acetaldeide 1/1000 di quello di etanolo – In presenza di isoforma con KM=1 mM, accumulo di acetaldeide – “Oriental flush” 0.9 0.8 0.7 0.6 0.5 0.4 0.3 0.2 0.1 0.0 0 60 50 [aldeide], µM [alcool] in sangue, g/l • Velocità: 10 g/h Diminuzione del tasso nel sangue: 0.15 g/l Indipendente dal tasso iniziale 40 30 KM=1 mM 20 KM=0.01 mM 10 1 2 3 4 5 6 7 8 Tempo, ore dopo 0.6 g/kg 9 10 0 0 1 2 3 4 5 6 7 8 9 10 Tempo, ore dopo 0.6 g/kg REGOLAZIONE DELL’ATTIVITÀ ENZIMATICA Necessità fisiologica; Catene enzìmatiche; Controllo del livello di enzima; Ubiquitina; Fattori fisici e chimici di modulazione dell’attività; Inibizione competitiva e non competitiva; Compartimentalizzazione intracellulare; Modificazioni allosteriche e covalenti; Allosterismo e cooperatività; Cinetica degli enzimi allosterici e cooperativi. 25 Il buon funzionamento della cellula richiede che le varie attività enzimatiche siano regolate • In condizioni di stato stazionario, i processi cellulari non funzionano mai al massimo delle loro capacità, ma possono anche venire completamente disattivati: – Non produrre prodotti inutili – Economizzare energia – Aumentare rapidamente l’attività se necessario Reversibilità delle reazioni enzimatiche • L’enzima favorisce il raggiungimento dell’equilibrio, quindi in teoria potrebbe catalizzare anche la reazione inversa A ⇐⇒ B • Ma, se B è substrato di una reazione successiva, la prima reazione diventa praticamente irreversibile A→B→C • Ciò vale per catene anche molto lunghe di reazioni (catene enzimatiche) A→B→C→D→E • Anche se le singole reazioni enzimatiche sono reversibili, in pratica il flusso è unidirezionale perché il prodotto di una reazione funge da substrato per la reazione successiva • Spesso il prodotto terminale di una sequenza di reazioni (E) inibisce il deflusso della stessa catena a livello della prima reazione A → B – L’accumulo di E “chiude il rubinetto” – La mancanza di E “riapre il rubinetto” A→B→C→D→E 26 Caratteristiche generali delle reazioni regolatorie • • • • ΔG<<0 (maggior efficacia) Precoci nella catena enzimatica Inibizione o regolazione da parte del prodotto finale Gli enzimi coinvolti sono spesso multimerici – Capacità catalitica = (concentrazione di enzima) x (efficienza catalitica) Controllo della concentrazione di enzima (regolazione lenta) Regola generale: Tutte le strutture cellulari ed extracellulari (ad eccezione dell’eritrocita umano) sono sottoposte a ricambio ed equilibrio dinamico Quindi, ad ogni istante, il livello di un enzima dipende da: • Velocità di sintesi della proteina • Velocità di trasformazione da zimogeno in enzima attivo • Velocità di degradazione 27 Velocità di sintesi • Può aumentare in seguito all’accumulo intracellulare del substrato (es: operon del lattosio) • Può aumentare in presenza di induttori gratuiti senza relazione col substrato • Può essere repressa dai prodotti terminali della catena enzimatica (es: alcuni aminoacidi (His, Leu), effetto glucosio in E.coli) Trasformazione da zimogeno (pro-enzima) in enzima attivo proinsulina → insulina 28 Chimotripsinogeno → chimotripsina Attivazione degli zimogeni pancreatici mediante taglio proteolitico 29 Velocità di degradazione Dipende da: • Lisosomi, pinocitosi, endocitosi • Ormoni (insulina, glucagone, glucocorticoidi…) • Ubiquitina (The kiss of death), Nobel Prize 2004 – Aaron Ciechanover, Avram Hershko e Irwin Rose, Medical Sciences, Technion in Haifa and UC Irvine College of Medicine in California) – Proteina altamente conservata (76 aa) e ubiquitaria – Attivata da ATP • Gli altri enzimi proteolitici non dipendono da ATP – Si lega covalentemente alla proteina-bersaglio (Lys) – Il complesso proteina-ubiquitina marcato per la distruzione Funzioni biologiche della proteolisi ubiquitinadipendente • Regolazione del ciclo cellulare – Essenziale per uscire dalla fase mitotica e separare i cromosomi durante mitosi e meiosi – Malfunzionamenti causano formazione di cellule con numero anomalo di cromosomi (Down e tumori solidi) • Riparazione del DNA, cancro e apoptosi – Essenziale per mantenere il livello di p53 (il “guardiano del genoma”) • Risposte immunologiche e infiammatorie – Regolazione di NF-kB (fattori di trascrizione) per attivare l’espressione genica – Generazione di peptidi MHC (difesa contro le infezioni virali) • Fibrosi cistica – Coinvolta nella mutazione di CFTR (transmembrane conductance regulator) 30 Compartimentalizzazione • Si può ottenere un forte aumento di concentrazione di molecole compartimentalizzandole in una zona delimitata da una membrana • Esempio: GLUcose Transporter (GLUT) – GLUT1, costitutivo – GLUT4, inducibile da insulina Modulazione dell’attività dell’enzima (regolazione veloce) • pH • Temperatura • Inibizione reversibile ed irreversibile: – Competitiva – Non-competitiva – Uncompetitiva • Modificazioni allosteriche • Modificazioni covalenti 31 pH, temperatura e attività enzimatica • Ogni enzima ha il suo pH ottimale Il pH ottimale può non corrispondere col pH in cui si trova l’enzima • Per ogni enzima, esiste una temperatura ottimale La temperatura ottimale può non corrispondere con la temperatura in cui si trova l’enzima (37°C) Aumento dovuto alla temperatura (1.7-2.5 ogni 10°C) Velocità • • Diminuzione dovuta alla denaturazione della proteina Risultato 0 10 20 30 40 50 Temperatura Inibizione competitiva e non-competitiva Competitiva: Substrato e inibitore si legano al sito attivo dell’enzima Il complesso enzima+substrato è inattivo Non-competitiva: Quando l’enzima lega l’inibitore, l’enzima cambia la conformazione e diventa inattivo 32 Inibizione competitiva • I simile a S, compete con S per il sito attivo di E • Inibizione reversibile all’aumentare di [S] • Vmax non cambia, KM aumenta v0 1/v0 Vmax ↑ inibitore → inibitore Vmax/2 1/Vmax → KM [S] → 1/KM 1/[S] Esempi di inibizione enzimatica competitiva CH3OH CH3-CH2OH Metanolo Etanolo 33 Esempi di inibizione enzimatica competitiva Metotrexato o antifolato, antileucemico che rallenta la biosintesi di purine e pirimidine Esempi di inibizione enzimatica competitiva Fluorouracile, analogo della mercaptopurina 34 Esempi di inibizione enzimatica competitiva Substrati suicidi o antimetaboliti, prevengono la reazione col substrato e/o disattivano l’enzima Penicillina inibisce transpeptidasi che stabilizza le membrane batteriche Inibizione non competitiva • • • • I reagisce su un sito diverso dal sito attivo Tende a disattivare E Inibizione non reversibile all’aumentare di [S] KM non cambia, Vmax diminuisce v0 1/v0 ↓ inibitore ↑ inibitore ↓Vmax ↑ 1/Vmax ↓Vmax/2 KM [S] 1/KM 1/[S] 35 Esempi di inibizione non-competitiva • Degradazione proteica (temperatura, estremi di pH) • Molti veleni e composti mercuriali (reagiscono con -SH dei residui di Cys) • Cianuro, reagisce con gli ioni metallici degli enzimi della catena respiratoria • Diisopropil fluorofosfato (Sarin), gas nervino, inibisce gli enzimi contenenti Ser (acetilcolinesterasi) Inibizione uncompetitiva I si lega al complesso ES 36 Inibizione uncompetitiva • L’inibitore reagisce con il complesso ES • Cambiamenti simultanei di KM e Vmax V0 1/V0 + inibitore Vmax + inibitore 1/Vmax KM [S] 1/KM 1/[S] Modificazioni covalenti degli enzimi • La fosforilazione AUMENTA l’attività di alcuni enzimi – Glicogeno fosforilasi, citrato liasi, fosforilasi b chinasi, HMG-CoA reduttasi chinasi e altri... • La fosforilazione DIMINUISCE l’attività di altri enzimi – Acetil-CoA carbossilasi, glicogeno sintasi, piruvato deidrogenasi, HMG-CoA reduttasi e altri... • The Nobel Prize in Physiology or Medicine 1992 – Edmond H. Fischer and Edwin G. Krebs, Professors Emeritus, University of Washington, Seattle, USA – … for their discoveries concerning reversible protein phosphorylation as a biological regulatory mechanism 37 cAMP Ormone cAMP come secondo messaggero Proteina G Recettore Adenilato ciclasi ATP cAMP R R R C R C Protein chinasi A inattiva C C Protein chinasi A attiva 38 Allosterismo allo = l’altro • • • • Attività enzimatica sotto il controllo di un modulatore Classe K o V: altera KM o Vmax Inibitore o stimolatore Interazione eterotropica: il modulatore è diverso dal substrato • Interazione omotropica: il modulatore funge anche da substrato Jacques Monod Nobel Prize 1965 Quando due leganti si legano alla stessa proteina, possono influenzare l’uno il legame dell’altro • Esochinasi (HK) Glucosio + ATP ⇐⇒ glucosio-6-fosfato + ADP • Quando HK lega glucosio, l’affinità per ATP aumenta • Quando HK lega ATP, l’affinità per glucosio aumenta 39 Allosterismo • Effetto reciproco di due leganti quando i siti di legame sono separati • Se i due leganti si legano alla STESSA conformazione di un enzima, ne risulta un’interazione POSITIVA • Se i due leganti si legano a conformazioni DIVERSE , ne risulta un’interazione NEGATIVA Modello di enzima allosterico multimerico A provoca un cambiamento conformazionale che facilita il legame con S Il cambiamento conformazionale è trasmesso alla subunità adiacente Allosterismo e cooperatività: – Allosterismo: cambiamenti conformazionali di una subunità in risposta ad un effettore allosterico – Cooperatività: cambiamenti conformazionali di una subunità in risposta al cambiamento conformazionale della subunità adiacente – Conformazione privilegiata nell’evoluzione naturale: il destino di una molecola è associato alla presenza/assenza di un’altra A S A S • • • A 40 Cinetiche degli enzimi allosterici e cooperativi • Sigmoide, non iperbolico • NON-Michaelis-Menten • Sempre più efficaci all’aumentare del numero di subunità v0 4 subunità 1 subunità 2 subunità [S] 41
Scarica