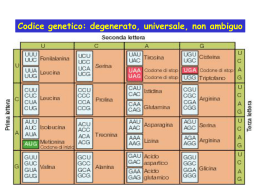
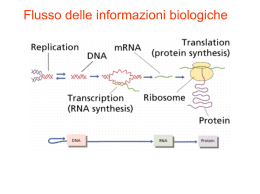
TRASCRIZIONE E MATURAZIONE DELL’RNA LA TRASCRIZIONE COME PRIMA TAPPA NEL FLUSSO DAL GENOTIPO AL FENOTIPO LA TRASCRIZIONE E’ IL PROCESSO ATTRAVERSO CUI SI PASSA DAL LINGUAGGIO DESOSSIRIBONUCLEOTIDICO DEL DNA A QUELLO RIBONUCLEOTIDICO DELL’RNA ATTRAVERSO LA TRASCRIZIONE NON VENGONO SINTETIZZATI SOLO GLI RNA CHE CODIFICANO PROTEINE (mRNA (mRNA), ), MA ANCHE SVARIATI ALTRI TIPI DI RNA (tRNA (tRNA;; rRNA rRNA;; E PICCOLI RNA NON CODIFICANTI QUALI: snRNA snRNA;; snoRNA snoRNA;; scRNA;; miRNA scRNA miRNA)) CHE SVOLGONO FUNZIONI CHIAVE NELLA FISIOLOGIA DELLA CELLULA. IN GENERALE IL PRODOTTO (RNA) DELLA TRASCRIZIONE PRENDE NOME DI TRASCRITTO TRASCRITTO.. L’INSIEME DI MOLECOLE DI RNA (CLASSICAMENTE mRNA mRNA)) ALL’INTERNO DI UN BEN PRECISO MOMENTO DELLA VITA DI UNA SPECIFICA CELLULA, COSTITUISCONO IL SUO TRASCRITTOMA. L’APPARATO ENZIMATICO CHE OPERA VIENE DEFINITO RNA POLIMERASI. POLIMERASI. UNIONE DI RIBONUCLEOSIDI MONOFOSFATO PER FORMARE UNA CATENA POLIRIBONUCLEOTIDICA. I PRECURSORI DELLA SINTESI SONO I NUCLEOSIDI TRIFOSFATO TRIFOSFATO.. L’ENERGIA CHE OCCORRE PER LA FORMAZIONE DEL LEGAME FOSFODIESTERICO È DATA DALL’ELIMINAZIONE DEL PIROFOSFATO PER IDROLISI DEL LEGAME. RNAn + rnPPP = RNA(n+1) + ppi SI INDICA COME PROMOTORE IL SITO DEL DNA OVE SI LEGA L’RNA POLIMERASI PRIMA DI INIZIARE LA TRASCRIZIONE! GENERALMENTE SOLO UNA DELLE DUE ELICHE DI DNA VIENE COPIATA COME RNA. IL FILAMENTO CHE FUNGE DA STAMPO E’ DETTO FILAMENTO SENSO,, MENTRE L’ALTRO FILAMENTO (CHE AVRA’ SEQUENZA IDENTICA SENSO ALL’RNA DI NEOSINTESI, A MENO DELLA T IN LUOGO DELL’U), E’ DETTO ANTISENSO (O NON SENSO). SENSO). UNO STESSO FILAMENTO PUO’ ESSERE SENSO IN ALCUNI TRATTI ED ANTISENSO IN ALTRI! LA DIREZIONE DI SINTESI DELL’RNA AVVIENE SEMPRE SECONDO L’ANDAMENTO 5’5’-3’! TRASCRIONE NEI BATTERI NEI BATTERI LA RNA POLIMERASI E’ COSTITUITA DA UN CORE DI 4 SUBUNITA’ (2 SUBUNITA’ α, UNA β ED UNA β’) [APOENZIMA [APOENZIMA], ], CUI, ALL’INIZIO DELLA TRASCRIZIONE, SI LEGA UNA QUINTA SUBUNITA’ ((σ σ), A FORMARE L’ L’OLOENZIMA OLOENZIMA.. SETs SPECIFICI DI PROMOTORI VENGONO RICONOSCIUTI DA SPECIFICHE SUBUNITA’ σ! STRUTTURA DEL PROMOTER PROCARIOTICO PRIBNOW BOX i) INIZIO: RNAINIZIO: RNA-POL (OLOENZIMA) SI LEGA ALLA REGIONE PROMOTER FACENDO SI CHE LA DOPPIA ELICA SI SEPARI. AVVIENE LA FORMAZIONE DELLA PRIMA COPPIA DI BASI TRA IL DESOSSIRIB. +1 DEL FILAMENTO STAMPO ED IL PRIMO RIBONUCL. DELLA CATENA DI RNA NASCENTE. QUESTA FASE NON NECESSITA DI ATP (AL CONTRARIO DEGLI EUCARIOTI); ii) ALLUNGAMENTO ALLUNGAMENTO:: LA SUBUNITA’ σ SI STACCA DALL’OLOENZIMA E L’OLOENZIMA CONTINUA LA SUA ATTIVITA’ DI SINTESI IN DIREZIONE 5’5’-3’; iii) FINE FINE:: PUO’ ESSERE ρDIPENDENTE O ρINDIPENDENTE. SPESSO GLI RNA (mRNA (mRNA;; tRNA ed rRNA rRNA)) DEI PROCARIOTI SONO POLICISTRONICI POLICISTRONICI,, CIOE’ ALL’INTERNO DI UN UNICO TRASCRITTO SONO CONTENUTE INFORMAZIONI PER LA CODIFICA DI PIU’ PROTEINE ((mRNA mRNA), ), PIU’ rRNA O PIU’ tRNA! tRNA! L’INTERA STRUTTURA GENICA POLICISTRONICA E’ DETTA OPERONE!! OPERONE TRASCRIONE NEGLI EUCARIOTI 3 RNARNA-POLIMERASI POLIMERASI,, COSTITUITE DA UN CORE ENZIMATICO E DA FATTORI GENERALI DI GTF.. I GTFs DELLA POLII TRASCRIZIONE O GTF SONO ALMENO 7: TFIIA TFIIA;; TFIIB TFIIB;; TFIID TFIID;; TFIIE TFIIE;; TFIIF;; TFIIH TFIIF TFIIH;; TFIIJ TFIIJ.. NOME LOCAL. CELL. TIPO RNA SENSIBILITA’ α-AMANITINA RNA POL I Nucleolo 45S (28S; 18S; 5.8S rRNA rRNA)) - RNA POL II Nucleoplasma mRNA; mRNA; snRNA; snRNA; snoRNA;; miRNA snoRNA +++ RNA POL III Nucleoplasma tRNA; tRNA; 5S rRNA; rRNA; scRNA;; alcuni snRNA scRNA + GENE EUCARIOTICO Promotore geni codificanti proteine! STRUTTURA DEL PROMOTER DEI GENI EUCARIOTICI CODIFICANTI PROTEINE NOTA:: ESISTONO NOTA ANCHE PROMOTERS TATA LESS TATA BOX BINDING PROTEIN TBP Saddle-like domain DNA BINDING TATA BOX TAF5 stabilizes TAFs interaction, interaction, specially histonehistonelike ones (TAF6, TAF9) TAF1: Acetyl transferase activity Interaction with TFIIF TAF12 TAF8 TAF7 TAF4 TAF10 TBP TAF6TAF11 TAF6 TAF11 TAF5 TAF5 TAF8 TAF3 TAF12 TAF11 TAF9TAF13 TAF10 TATA BOX TAF6 TAF9 TAF13 TAF4 TAF3 DNA BENDING TFIID HETEROTETRAMER (RAP30)2 (RAP74) 2 (RAP30)2 BINDS RNA POL II AND TFIIB. RAP74 INTERACTS WITH DNA. TFIIF STABILIZES THE INTERACTION OF RNA POLII WITH PROMOTER AND STIMULATES ELONGATION RATE DAB TWO DOMAIN: HETEROTRIMER N-TERMINAL (A, B, C SUBUNITS) BINDS PROMOTER REGION WITH TFIID ZnZn-RIBBON AND TFIIA TFIIF TFIID CORE DOMAIN TFIIE TFIIB TFIIH TFIIE MODULATES THE HELICASE AND KINASE ACTIVITIESOF TFIIH TFIIB INTERACTS WITH SADDLESADDLE -LIKE DOMAIN OF TBP AND WITH BENDED DNA ON SIDES OF TATA BOX. TBP/TFIIB COMPLEX RECRUITS RNA POLII AND OTHER GTF P BY STIMULATING CTD DOMAIN RNA POLII PHOSPHORYLATION TFIIH STIMULATES PROMOTER PHOSPHORYLATION OF MELTING AND IT RNA POLII CTD HAS KINASESTIMULATES PROMOTER ACTIVITY MELTING AND AGAINST RNA POL II TRANSCRIPTION START ENHANCER È IL TERMINE USATO PER DEFINIRE SPECIFICHE SEQUENZE DI DNA IN GRADO DI AUMENTARE L'EFFICACIA DEI PROMOTORI NELL'ATTIVAZIONE DELLA TRASCRIZIONE. TRASCRIZIONE. GLI ENHANCER SVOLGONO IL LORO RUOLO ATTRAVERSO L'ASSOCIAZIONE CON DIVERSE PROTEINE, TRA CUI DIVERSI FATTORI COINVOLTI NELL'AVVIO DELLA TRASCRIZIONE STESSA. NEI GENI DEGLI EUCARIOTI GLI ENHANCERS POSSONO DISTARE DALLA REGIONE CODIFICANTE ANCHE PIÙ DI 50 KB. KB. IL SILENCER È UNA SEQUENZA DI DNA IN GRADO DI LEGARE DEI FATTORI DI TRASCRIZIONE DETTI REPRESSORI. QUANDO IL SILENCER È LEGATO DAL REPRESSORE, L’RNA POLIMERASI NON È IN GRADO DI INIZIARE LA TRASCRIZIONE. TERMINAZIONE DELLA TRASCRIZIONE NEI GENI EUCARIOTICI MATURAZIONE DELL’mRNA DELL’mRNA i) CAPPING ii) METILAZIONE iii) POLIADENILAZIONE iv) SPLICING v) EDITING i) 5’5’- CAPPING 5’ capping 7’methyl guanylate Legame 5’ – 5’ Metilazione del 2’C PRINCIPALI FUNZIONI DEL 5’5’- CAPPING PROTEZIONE E STABILIZZAZIONE DEGLI mRNA, TRASPORTO DAL NUCLEO AL CITOSOL, INIZIO DELLA TRADUZIONE. ii) ii) POLIADENILAZIONE QUESTA SEQUENZA SI TROVA A CA. CA. -10/10/-35 RISPETTO AL SITO DI POLIADENILAZIONE NOTA:: ALCUNI hnRNA (Es.: NOTA QUELLI DEI GENI CODIFICANTI GLI ISTONI) NON HANNO UNA CODA DI POLIPOLI-A! DAPPRIMA I CLEAVAGE FACTORS TAGLIANO A 10/35 NT. A VALLE DELLA SEQUENZA AAUAAA, QUINDI I CPF (CLEAVAGE AND POLYADENILATION FACTORS) CONSENTONO ALLA POLI(A) POLIMERASI DI RICONOSCERE LA SEQ. SEQ. AAUAAA E DI ADD. LE “A” ! PRINCIPALI FUNZIONI DELLA POLIADENILAZIONE LA CODA DI POLI A CONTRIBUISCE IN MODO MOLTO IMPORTANTE ALLA STABILITA’ DEGLI mRNA ED ALL’INCREMENTO DELLA EFFICIENZA TRADUZIONALE! iii) iii) SPLICING PROCESSO ATTRAVERSO CUI VENGONO ELIMINATI GLI INTRONI. IL COMPLESSO RIBONUCLEOPROTEICO (FATTO DA snRNA + PROTEINE, A FORMARE LE snRNPs) snRNPs) ADIBITO AL CONTROLLO DELLO SPLICING PRENDE NOME DI SPLICEOSOMA SPLICEOSOMA!! LA RIMOZIONE DI UN INTRONE AVVIENE ATTRAVERSO DUE REAZIONI SEQUENZIALI DI TRASFERIMENTO DI FOSFATO, NOTE COME TRANSESTERIFICAZIONI. QUESTE UNISCONO DUE ESONI RIMUOVENDO L’INTRONE COME UN “CAPPIO” L’ELIMINAZIONE DEGLI INTRONI PREVEDE LA FORMAZIONE DI UNA STRUTTURA A CAPPIO DETTA “LARIAT “LARIAT”. ”. Lo splicing avviene ad opera dello spliceosoma spliceosoma,, costituito da snRNPs (small nuclear ribonucleoprotein particles) formati da snRNA e proteine. proteine. SPLICING ALTERNATIVO PERMETTE LA FORMAZIONE DI TRASCRITTI MATURI DIVERSI A PARTIRE DA UNO STESSO hnRNA cell 2 cell 1 (4 exons exons)) tissue specific ALTRI MECCANISMI CHE PRODUCONO FORME ALTERNATIVE DI TRASCRITTI, PARTENDO DALL’INFORMAZIONE CONTENUTA IN UNO STESSO GENE SONO: i) RICONOSCIMENTO DI SITI DI INIZIO DELLA TRASCRIZIONE ALTERNATIVA (Es.: GENE PER IL GnRH - Gonadotropin Releasing Hormone Hormone- CHE PRESENTA DUE TRASCRITTI DIVERSI, UNO A LIVELLO IPOTALAMICO ED UNO A LIVELLO DI PLACENTA ED ALTRI TESSUTI. IL SECONDO SITO DI INIZIO SI TROVA A -579 E DIPENDE DA UN SECONDO PROMOTORE); ii) ii) SEGNALI DI TERMINAZIONE DELLA TRASCRIZIONE ALTERNATIVI STRUTTURA DELL’ hnRNA Cap 5’ 7mGppp initiation 5’ untranslated region AUG translated region UGA 3’ untranslated region termination polyadenylation signal AAUAAA (A)~200 3’ poly(A) tail • all mRNAs have a 5’ cap and all mRNAs (with the exception of the histone mRNAs) contain a poly(A) tail • the 5’ cap and 3’ poly(A) tail prevent mRNA degradation • loss of the cap and poly(A) tail results in mRNA degradation MATURAZIONE DEGLI rRNA E tRNA I GENI PER GLI rRNA 18S, 5.8S E 28S IN H. sapiens SONO RAGGRUPPATI IN CLUSTER. TALI UNITA’ SONO RIPETUTE IN TANDEM CIRCA 250 VOLTE E DISTRIBUITE SU 10 ANSE CROMOSOMICHE LOCALIZZATE SUI BRACCI CORTI DEI CHR. ACROCENTRICI 13, 14, 15, 21 E 22. I GENI PER I tRNA SONO ORGANIZZATI IN GRUPPI E, IN CIASCUN CLUSTER, CIASCUN GENE E’ SEPARATO DAI GENI VICINI DA SPAZIATORI INTERGENICI. IL PREPRE-tRNA DEVE PERDERE ALCUNE SEQ. SEQ. AL 5’ E 3’ E, IN ALCUNE SPECIE DEVE AVVENIRE ANCHE UN SELFSELFSPLICING DI UN INTRONE. ALTRI EVENTI MATURATIVI RIGUARDANO ESSENZIALMENTE LA MODIFICA DI ALCUNE BASI E L’AGGIUNTA AL 3’ DELLA SEQUENZA 5’5’-CCA CCA-3’ AD OPERA DELLA tRNA NUCLEOTIDIL TRANSFERASI.
Scarica