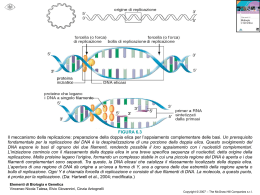
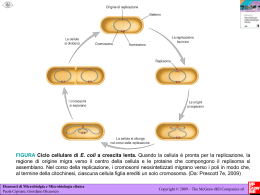
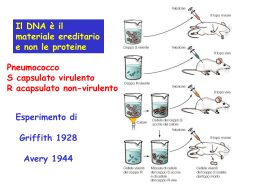
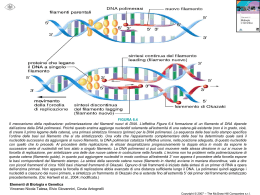
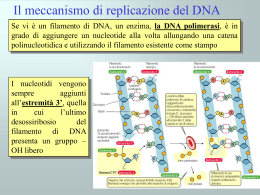
Regolazione della replicazione del DNA durante il ciclo cellulare Le cellule eucariote durante lo sviluppo e la divisione duplicano il loro genoma in modo molto fedele. La precisione di questo processo dipende in larga misura da meccanismi di regolazione che accoppiano la replicazione del DNA alla progressione del ciclo cellulare. Per duplicare in modo efficiente il genoma (da 107 a più di 109 bp organizzato in cromosomi) gli organismi eucarioti hanno sviluppato, durante l’evoluzione, un meccanismo che prevede l’inizio della replicazione del DNA a livello di siti multipli sul DNA cromosomale. Questo meccanismo è finemente regolato ed in particolare la replicazione viene innescata in un preciso stadio del ciclo cellulare, a livello di centinaia se non di migliaia di siti specifici all’interno dei cromosomi parentali, ed, inoltre, l’inizio della replicazione deve essere impedita sugli stessi siti contenuti nel DNA di nuova formazione delle cellule figlie. Questo meccanismo assicura che ogni segmento del genoma sia duplicato una sola volta durante il ciclo cellulare. Inoltre, le cellule eucariote hanno sviluppato dei controlli supplementari, chiamati checkpoint, che alterano il normale avanzamento del ciclo cellulare in risposta ad eventi potenzialmente genotossici come il danno al DNA o anomalie nella sintesi dello stesso. Questi ed altri meccanismi regolatori collaborano insieme per mantenere l’integrità del genoma durante il processo di regolazione. Negli ultimi 15 anni passi da gigante sono stati fatti nella comprensione dei meccanismi che portano alla replicazione del DNA. Grazie agli studi compiuti su semplici sistemi modello come Saccharomyces cerevisiae, Schizosaccharomyces pombe e Xenopus leavis è stato possibile caratterizzare le proteine che agiscono all’origine di replicazione, responsabili dell’inizio della REGOLAZIONE DELLA REPLICAZIONE DEL DNA DURANTE IL CICLO CELLULARE Primi studi Cellule umane a vari stadi del ciclo cellulare vengono fuse per formare un’unica cellula formata da 2 nuclei ORIGINE DI REPLICAZIONE • Sequenza di DNA alla quale si lega il complesso proteico che promuove la replicazione del DNA • Origine di replicazione più conosciuta è quella del Saccaromices cervisiae (ARS) • ARS è costituita da corte sequenze distribuite in 100-200 bp: - elemento A →recluta primo complesso proteico di replicazione ORC (11 bp ricchi in AT) - elementi B →B1 B2 B3 B1 recluta ORC ORC Origin Replication Complex • • • • • Complesso proteico formato da 6 proteine (Orc1-6) in S. cerevisiae Lega per primo origine di replicazione Orc1 Orc4 Orc6 possiedono sito AAA+ contenente ATP-binding domain responsabile dell’assemblaggio e rimodellamento del complesso ORC identificato in organismi superiori Recluta fattore di replicazione CDC6 REPLICAZIONE DEL DNA CDC6/CDC18 Promuove l’inizio della replicazione del DNA MCM Complesso proteico costituito da 6 Componenti (Mcm2-Mcm7) caratterizzato da attività elicasica ed elongazione della replicazione Caratteristiche molecolari: • Sito AAA+ →ATP-binding domain • Sito di interazione con CDK CDC45 Lega complesso d’inizio dopo l’eliminazione di Cdc6 Responsabile del reclutamento della DNA polimerasiα/primasi REGOLAZIONE POSITIVA DELLA REPLICAZIONE DEL DNA • ORC CDC6/CDC18 e MCM appartengono alla famiglia delle proteine AAA+ con attività ATPasica, queste sono caratterizzate da una regione conservata che contiene – il sito di legame all’ ATP responsabile del cambiamento conformazionale che media la loro interazione con i diversi complessi proteici, attraverso un meccanismo a cascata. – Un piccolo dominio di 3 alfa eliche definito AAA+ che è caratteristico di questo gruppo di proteine. • In CDC6/CDC18 e MCM contengono inoltre una ulteriore regione conservata che interagisce con CDK e contiene siti di fosforilazione CDK – dipendenti • Cdc7-Dbf4 kinases Cdc7 è una serina/treonina chinasi la cui attività è essenziale per la formazione di un attivo complesso di pre-inizio della replicazione. L’attività di Cdc7 è mediata dalla proteina Dbf4 la quale sembra indirizzi Cdc7 verso il complesso di preiniziazione. REGOLAZIONE DELLA REPLICAZIONE: ad ogni ciclo cellulare si ha una sola replicazione del DNA CDK Ciclin-dependent kinases Famiglia di proteine la cui funzione catalitica è attivata da cicline Promuovono l’inizio della replicazione e prevengono fenomeni di re-iniziazione della replicazione durante lo stesso ciclo cellulare CDC6/CDC18 Fosforilazione dei CDK-phosphorilation sites contribuisce all’inibizione del fenomeno di re-iniziazione della replicazione promuovendo il distacco della proteina dal complesso ORC, l’esporto dal nucleo, ubiquitinazione e la successiva eliminazione mediante proteaosoma Il distacco di Cdc6 favorisce il legame di Cdc45 al complesso d’inizio e quindi l’elongazione della replicazione MCM L’attivita’ delle CDK (Cdk2) durante le fasi G2 ed M del ciclo cellulare previene l’associazione delle proteine Mcm alla cromatina inibendo la formazione del complesso d’inizio della replicazione nuova sintesi di DNA. Sebbene non siano stati ancora svelati tutti i dettagli molecolari che caratterizzano il pathway dell’iniziazione della replicazione, è chiaro che un unico set proteico di inizio si assembla sull’origine di replicazione di tutti gli organismi eucarioti e che le attività di queste proteine sono regolate da specifiche proteine chinasi. ORIGINE DI REPLICAZIONE La replicazione dei cromosomi viene innescata a partire da precise sequenze di DNA, conosciute come ORIGINE DI REPLICAZIONE. Negli organismi eucarioti le sequenze più caratterizzate sono quelle presenti nei S. cerevisiae, le ARS (autonomously replicating sequences), le quali sono costituite da diverse sotto-sequenze molto conservate di circa 100-200 bp, chiamate ELEMENTI A ed ELEMENTI B. La sotto-sequenza più importante è l’elemento A formato da un dominio di 11 bp ricco in AT, il quale è essenziale per il legame delle proteine ORC (origin recognition complex) che costituiscono il primo complesso proteico necessario per l’inizio della replicazione. L’elemento B è costituito da 3 domini, B1 B2 e B3 che rispettivamente hanno le funzione di rappresentare il secondo sito di legame per le proteine ORC, aprire la molecola di DNA e stimolare il processo di iniziazione. Le origini di replicazione degli organismi eucarioti diversi da S. cerevisiae sono strutturalmente molto simili anche se salendo lungo la scala evolutiva, fino ad arrivare all’uomo, la loro complessità aumenta. COMPLESSO PROTEICO DI INIZIAZIONE L’iniziazione della replicazione nelle cellule eucariote è un processo che si sviluppa in due diversi steps. In fase G1 la cellula diventa competente all’iniziazione della replicazione grazie al legame sull’origine di replicazione delle diverse proteine che compongono il complesso. Grazie all’azione di specifiche proteine chinasi CDKs (cyclin-dependent kinases) ed a proteine chinasi della famiglia delle cdc7, il complesso ormai completamente assemblato viene attivato favorendo, così, l’inizio della sintesi del DNA, durante la fase S. Oltre all’innesco della replicazione del DNA, le CDKs impediscono il ri-assemblaggio delle proteine sull’origine di replicazione. Così, grazie all’azione delle CDKs la cellula inizia la sintesi di DNA e simultaneamente perde la competenza ad un secondo ciclo di replicazione. La competenza è ripristinata attraverso l’eliminazione dell’attività delle CDKs durante la fase M del ciclo cellulare. Origin recognition complex (ORC) Il complesso proteico ORC gioca un ruolo centrale nell’iniziazione della replicazione del DNA poiché per primo lega l’origine di replicazione ed inoltre recluta gli altri complessi importanti per questo meccanismo. L’ORC più caratterizzato è quello appartenente a S. cerevisiae il quale è costituito da 6 proteine (ScORC1-6) che sono conservate anche in organismi superiori al lievito come Drosophila e uomo; questo suggerisce che il meccanismo di inizio replicazione sia molto simile nei diversi eucarioti. La formazione di un ORC stabile richiede ATP che una volta reclutata viene idrolizzata dal complesso condizionandone il legame con gli altri fattori di iniziazione. Cdc6 Fattore proteico, altamente conservato nei diversi organismi eucarioti, che si lega ad ORC ed è essenziale per il processo di iniziazione della replicazione ma non per la vera e propria sintesi di DNA. Cdc6 è una proteina ATPasi caratterizzata da una regione conservata AAA+ che contiene 2 domini: ATP-binding domain e una piccola sequenza composta da tre “helix bundle”. Il legame e l’idrolisi dell’ATP regola le conformazione della proteina influenzando la stabilità del complesso di iniziazione. Queste 2 domini sono presenti anche in altre proteine coinvote nel processo di iniziazione della replicazione del DNA come ORC e MCM. Oltre alla regione AAA+, Cd6 contiene all’estremità N-terminale un sito di fosforilazione dipendente da CDK. E’ stato osservato sia in S. cerevisiae che in S. pombe che una overespressione di questa proteina è sufficiente ad innescare passaggi multipli di replicazione di DNA all’interno dello stesso ciclo cellulare. Cdt1 Proteina altamente conservata nei vari organismi eucarioti che, come Cdc6, lega il complesso ORC. La sua struttura molecolare comprende diversi siti di fosforilazione dipendenti da CDK ed inotre è caratterizzata da 2 regioni con conformazione ad α-elica che si presume siano importanti per l’ interazione con altre proteine. La sua espressione è molto alta durante la fase G1, mentre durate la fase G2 e S si abbassa notevolmente. COMPLESSO MCM (Minichromosome Maintenance Proteins) Le MCM sono una famiglia di 6 proteine (da mcm2 a mcm7) la cui funzione nel processo di replicazione del DNA è duplice. • Le MCM, in presenza di Cdc6 e Cdt1, si legano all’origine di replicazione, dove prendono parte al complesso di iniziazione. • La presenza di questo complesso è stata riscontrata anche in siti progressivamente più distanti da quello dell’origine di replicazione, correlabili alla posizione nel tempo della DNA polimerasi ε, suggerendo che MCM possa avere un ruolo anche a livello della forcella di replicazione. Studi biochimici, infatti, hanno confermato come il complesso MCM sia necessario per l’elongazione della nuova molecola di DNA in quanto caratterizzato da proprietà elicasiche (mcm4-mcm5mcm6). CdC45 La proteina Cdc45 si associa alla all’origine di replicazione del DNA durante la fase G1 del ciclo cellulare. Come per MCM, la sua presenza viene anche riscontrata in sito lontani da quelli dell’origine di replicazione, suggerendo che Cdc45 abbia un ruolo importante nella forcella di replicazione. Questa proteina, infine, è coinvolta nel processo di reclutamento della DNA polimerasi α/ primasi sul complesso di iniziazione. REGOLAZIONE DEL COMPLESSO DI INIZIAZIONE CDK e Cdc7-Dbf4 sono proteine chinasi importanti per la regolazione del processo di iniziazione della replicazione nelle cellule eucariote, ed agiscono in differenti momenti e su differenti substrati. CDK, inoltre, ha un ruolo di regolazione negativa su tale meccanismo, bloccando la re-iniziazione della replicazione del DNA. CDK(cyclin-dependent Kinases) Le proteine CDK sono una famiglia di proteine chinasi che per svolgere la loro funzione catalitica hanno bisogno dell’interazione con proteine cicline attivanti. Grazie alla fosforilazione di siti specifici sui substrati proteici mediano il processo della replicazione. Cdc7-Dfb4 kinase Cdc7 è una proteina serina-treonina chinasi espressa a livelli costanti attraverso tutte le fasi del ciclo cellulare, la cui attività è influenzata dal fattore proteico Dfb-4, che si lega ad essa. L’attività di Cdc7 è bassa durante la fase G1 , aumenta durante la transizione G1/S, rimane alta durante tutta la fase G2 perpoi abbassarsi durante la fase M. INIZIAZIONE DELLA REPLICAZIONE E SUA REGOLAZIONE All’origine di replicazione è legato costitutivamente il complesso ORC al quale si legano le proteine Cdc6 e Cdt1. Durante la fase G1, Cdc6 e Cdt1 reclutano il complesso MCM; queste 3 componenti formano il complesso di pre- replicazione (pre-RC). La fase S è poi innescata dalle proteine CDK e Cdc7 che fosforilando siti specifici di Cdc6 e MCM attivano il complesso pre-RC. La fosforilazione di Cdc6, ad opera di Cdc7-Dbf4 provoca un cambiamento conformazionale della proteina che conduce al suo successivo distacco dal complesso e al legame di Cdc45. In modo analogo, la fosforilazione di MCM produce il suo distacco dal complesso durante il processo di sintesi del DNA. Una volta che la replicazione è stata completata MCM rimane nel nucleo come complesso solubile durante le fasi G2 e M. Il meccanismo che porta alla sintesi di una sola molecola di DNA per ogni ciclo cellulare coinvolge le proteine CDK e l’inattivazione dei fattori che compongono il complesso di pre-RC. La fosforilazione di Cdc6 provoca il distacco dal complesso della proteina, la sua successiva esportazione nel citoplasma e alla degradazione per mezzo del proteosoma. Anche la fosforilazione di Cdt1 comporta la distruzione della proteina, ed in caso di mancata degradazione Cdt1 viene inibita da un’altra proteina , la Geminina, la quale è espressa durante le fasi S, G2 e mitosi. Durante la mitosi le condizioni del complesso MCM cambiano profondamente. La rottura della membrana nucleare permette Cdc6 e Cdt1, non legato alla geminina, di legarsi al complesso. Questo processo permette il reclutamento di MCM che lega la cromatina di entrambe le cellule figlie.
Scarica