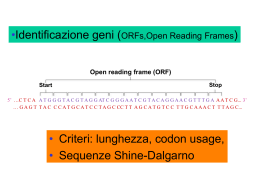
Bioinformatica Marin Vargas, Sergio Paul 2013 Wikipedia: La bioinformatica è una disciplina scientifica dedicata alla risoluzione di problemi biologici a livello molecolare con metodi informatici. La bioinformatica è la disciplina scientifica che cerca di risolvere problemi biologici mediante l’elaborazione informatica dell’informazione proveniente diretta o indirettamente da essere viventi. Tipi di informazione: Sequenze genomiche (DNA genomico: genomi, esomi o alcune regioni particolari del genoma). Sequenze proteiche (cDNA cioè DNA retrotrascritto a partire da un mRNA). Strutture 3D di proteine (NMR, Cristallografia), biologia strutturale. Immagini (RX, TAC, MRI, US, ecc). Concentrazioni di particelle nel sangue. Informazione di interazione tra molecole (systems biology). Informazione evoluzionistica. Pulsazioni, respiri, battiti cardiaci, ecc... La genomica è una branca della biologia molecolare che si occupa dello studio del genoma degli organismi viventi. In particolare si occupa della struttura, contenuto, funzione ed evoluzione del genoma. È una scienza che si basa sulla bioinformatica per l'elaborazione e la visualizzazione dell'enorme quantità di dati che produce. Estrazione e/o cattura di DNA da essere viventi. Sequenziamento del DNA con tecniche all’avanguardia come NGS (Next Generation Sequencing). Assemblaggio di genomi a partire da milioni di frammenti di DNA. Ri-sequenziamento di genomi. Allineamento di frammenti di DNA a un genoma di riferimento. Annotazione di genomi. Annotazione funzionale di geni all’interno di un genoma. Analisi di espressione genica mediante sequenziamento dei trascritti (RNA-Seq). GWAS (Genome Wide Association Studies). Analisi di varianti tra genomi (Variant calling o Chiamata delle varianti). … Ottimizzazione del protocollo bioinformatico per l’annotazione di geni codificanti proteine in genomi complessi Marin Vargas, Sergio Paul 2012 Con l’avvento del sequenziamento NGS a costi sempre più contenuti, il numero di genomi sequenziati si sta incrementando considerevolmente. Lo scopo di conoscere la sequenza genomica è principalmente indirizzato a capire la funzionalità dei geni. In passato l’annotazione di un genoma era molto dispendiosa. Oggi con le nuove tecnologie, è diventata alla portata di un singolo laboratorio. Rimane comunque un compito molto impegnativo. Annotare un genoma significa conoscere la localizzazione, la struttura e la funzionalità di tutti gli elementi che compongono l’intero genoma: • Geni codificanti proteine • Geni non codificanti proteine • Elementi regolatori • Elementi ripetuti • Pseudogeni • Altri elementi L’annotazione dei geni codificanti proteine, viene suddivisa in: Annotazione funzionale, consiste nel caratterizzare ogni singolo gene, assegnando una funzione biologica a ogni proteina codificata dal gene stesso. Annotazione genica o semplicemente annotazione, consiste nel definire all’interno del genoma: • La localizzazione di ciascun gene. • La struttura di ciascun gene (esoni, CDS, UTR). • Gli eventuali trascritti alternativi. Cap 5’ 3’ Poly-A AAAAAA mRNA maturo UTR CDS UTR 5’ 3’ Esone 1 3’ Esone 2 Esone 3 5’ DNA ATG STOP! Un gene codificante proteine è composto da diversi elementi: Esone: regione che viene mantenuta dopo la maturazione. Introne: regione che viene eliminata durante la maturazione. mRNA: RNA maturo, composto da esoni. CDS: regione codificante dell‘mRNA. UTR: regione non tradotta dell’mRNA. Metodi basati sull’allineamento delle evidenze sperimentali. Metodi basati sulla predizione genica ab initio. Metodi basati sulla predizione genica ab initio guidata da evidenze sperimentali. Metodi basati sul confronto tra genomi. 5 Si possono utilizzare diverse evidenze sperimentali, che opportunamente elaborate e allineate al genoma permettono di identificare le regioni codificanti proteine: • cDNA full-length: sequenze di RNA maturi (mRNA) retrotrascritti a cDNA, quindi completo di UTR e CDS. • EST (Expressed Sequence Tags): brevi frammenti parziali, tra 400-800 bp, di mRNA retrotrascritti a cDNA. • Proteine omologhe: sequenze aminoacidiche corrispondenti a proteine omologhe di organismi evolutivamente vicini. • Tiling arrays: microarray con sonde equamente spaziate su tutto il genoma, permettono l’identificazione di regione espresse mediante l’ibridazione di campione marcati. • MPSS: Massively Parallel Signature Sequencing, piattaforma che analizza il livello di espressione e identifica una regione di 17-20 bp degli mRNA tramite sequenziamento. • RNA-seq: frammenti di cDNA di lunghezza tra 50-150 bp che derivano dal sequenziamento shotgun di un intero trascrittoma. Sono dei brevi frammenti di lunghezza tra 400-800 bp di cDNA ottenuto dalla retrotrascrizione di un frammento di RNA maturo. Dalla sequenza proteica delle proteine si può risalire alla sequenza nucleotidica e quindi alla zona codificante (CDS) del gene che l’ha codificata. Sono sequenze di lughezza tra 50-150 bp che derivano dal sequenziamento shotgun di un intero trascrittoma, cioè dalla retro-trascrizione di tutto l’RNA in cDNA di un particolare momento cellulare, poi spezzato e sequenziato con tecnologie NGS. Per identificare le regioni codificanti i predittori utilizzano algoritmi e modelli matematici specifici che utilizzando informazione intrinseca dell’organismo analizzato cercano di identificare la localizzazione e la struttura dei geni. Sensori di segnale (signal sensors): permettono di identificare le giunzioni esone-introne e le estremità delle regioni codificanti. Sensori di contenuto (content sensors): permettono di identificare le regioni codificanti di lunghezza variabile. I predittori hanno bisogno di dati di esempio per imparare le caratteristiche dell’organismo analizzato (dati di training) e dei dati di prova per valutare l’accuratezza delle predizioni (dati di test). Predittore Augustus Snap GeneMark-ES GeneID FGenesh Genescan MZEF mGene.NGS Contrast GrailExp TwinScan/N-Scan Training in Utilizzo di Predizione di Utilizzo di Predizione Predizione locale per EST e Predizione geni RNA-Seq per dei trascritti degli UTR ab initio nuovi Proteine per la predizione alternativi eucarioti genomi la predizione SI SI SI SI SI SI SI SI SI SI SI SI SI SI SI SI SI SI SI SI SI SI SI SI NO SI SI NO NO SI SI NO SI SI NO NO SI NO SI NO SI SI SI SI SI NO NO SI NO SI NO SI NO NO NO SI NO NO SI NO SI NO SI SI SI NO SI NO NO SI NO NO NO NO NO NO SI Predizione genica ab initio: utilizza dati di training che potrebbero non essere rappresentativi di tutti i geni del genoma. Evidenze sperimentali: non coprono mai tutto il genoma, quindi non permettono l’annotazione completa di tutti i geni codificanti proteine. I migliori metodi di predizione genica utilizzano una metodologia ibrida tra predizione genica ab initio e l’utilizzo degli allineamenti delle evidenze sperimentali: cDNA EST Proteine RNA-Seq Creazione di un consensus utilizzando le evidenze sperimentali e le predizioni geniche. Ciascuna evidenza viene pesata dando un peso maggiore ai dati sperimentali rispetto alle predizioni. Principali programmi di integrazione: • Evidence Modeller • JIGSAW • GAZE Basate su automazione di programmi di predizione e allineamento esistenti. Vantaggio: relativamente semplici da utilizzare. Svantaggio: consentono un controllo limitato dei passaggi intermedi dell’annotazione. Pipeline di annotazione più utilizzate: • PASA • MAKER L’ottimizzazione del protocollo bioinformatico per l’annotazione dei geni codificanti proteine in genomi complessi. A questo scopo non verrà utilizzata una pipeline automatica di annotazione ma, attraverso la scelta di metriche adeguate, verrà valutato ogni singolo passaggio intermedio dell’annotazione in modo da fornire una procedura ottimizzata sulla base delle evidenze sperimentali a disposizione. Genoma dell’organismo eucariote Vitis vinifera, versione V1 PN40024 12X del consorzio French-Italian Public Consortium for Grapevine Genome, con una dimensione di 487 Mb. Motivi di questa scelta: • Il genoma è disponibile. • Ci sono dati sperimentali disponibili (EST, 454, RNA-Seq, cDNA full-length). 16.054 contig di cDNA full-length prodotte dal consorzio FrenchItalian Public Consortium for Grapevine Genome 3752 cDNA non ridondanti. Rimozione delle sequenze con ORF non completa 3.436 sequenze. Le 3.436 sequenze sono state suddivise in due gruppi in maniera del tutto casuale: • 936 sequenze di cDNA full-length training. • 2.500 sequenze di cDNA full-length test. EST: • 2.713.343 sequenze EST pubbliche (NCBI, Sequenziamento 454 + banca dati del consorzio). • Allineamento e generazione modelli genici con Gmap. • 1.649.082 trascritti putativi ridondati (56.630 non ridondanti). Proteine omologhe: • Allineamento al genoma delle sequenze proteiche di tutto il database SWISSPROT utilizzando Blat, Blast e Genewise. • 22.355 trascritti putativi ridondanti (5.808 non ridondanti). RNA-seq: • 114.726.580 reads RNA-seq sequenziati dal laboratorio di genomica dell’Università di Verona (pool di 45 campioni provenienti da 15 tessuti e organi a diversi stadi di sviluppo). • Allineamento e generazione modelli genici con suite Bowtie + Tophat + Cufflinks. • 40.324 trascritti putativi ridondanti (17.444 non ridondanti). Statistiche generali degli allineamenti delle evidenze sperimentali Statistiche generali Numero di modelli genici allineati Numero di modelli genici multi esonici Media della lunghezza dei modelli genici N50 della lunghezza dei modelli genici Media del numero di esoni per modello genico EST 56.630 19.485 1.034,12 2.257 3,30 Proteine omologhe 5.808 3.175 874,42 1.563 4,39 RNA-seq 17.444 17.366 2.236,89 2.751 6,75 Distribuzione della percentuale di sovrapposizione di nucleotidi tra allineamenti e riferimento Ho scelto i seguenti programmi di predizione genica, nei quali è stato realizzato il training con dati sperimentali di Vitis vinifera: • Augustus: supporta suggerimenti da evidenze sperimentali. • GeneID: supporta suggerimenti da evidenze sperimentali. • SNAP: realizza solo predizione ab initio. Sono state realizzate le seguenti predizioni: • Augustus ab initio • GeneID ab initio • SNAP ab initio • Augustus con suggerimenti RNA-seq • GeneID con suggerimenti RNA-seq I risultati delle predizioni sono state filtrati secondo: • • Eliminazione di tutte le predizioni di geni monoesonici (predizioni meno affidabili rispetto alle predizioni di geni multiesonici). Eliminazione di tutte le predizioni di geni con lunghezza della regione esonica inferiore a 200 basi. Statistiche generali delle predizioni ab initio Statistiche generali Numero di geni predetti Media della lunghezza dei geni N50 della lunghezza dei geni Media del numero di esoni per gene Augustus ab initio 30.510 1.122,73 1.455 4,44 GeneID ab initio 48.751 977,81 1.386 4,34 SNAP ab initio 64.431 1.020,27 1.563 6,14 Distribuzione della percentuale di sovrapposizione tra predizioni e riferimento Statistiche generali delle predizioni guidate da evidenze sperimentali Statistiche generali Numero di geni predetti Media della lunghezza dei geni N50 della lunghezza dei geni Media del numero di esoni per gene Augustus con RNA-seq 26.694 1.134,61 1.437 4,74 GeneID con RNA-seq 52.245 1.060,43 1.536 4,30 Distribuzione della percentuale di sovrapposizione di nucleotidi tra predizioni e riferimento Le statistiche generali da sole non consentono di valutare adeguatamente le differenze tra le predizioni, si rende quindi necessario fare una valutazione quantitativa dell’accuratezza. Sensibilità (SN) ed Specificità (SP): • Sensibilità misura quanto il predittore è in grado di fare predizioni. • Specificità misura quanto il predittore predice in modo corretto. Accuratezza (AC): • AC = (SN + SP) / 2 Tre livelli d’indagine: • Locus genico: misura la capacità di rilevare la presenza di un locus. • Regioni esoniche, misura la capacità di distinguere tra esoni e introni. • Giunzioni esone-introne, misura la capacità di predire in maniera corretta la struttura dei geni. Confronto degli allineamenti e delle predizioni contro il dataset di riferimento (loci genici) Evidenze Sensibilità Specificità Accuratezza EST 0,5680 0,6428 0,6054 Proteine omologhe 0,1872 0,6047 0,3960 RNA-seq 0,6140 0,7362 0,6751 Augustus ab initio 0,4612 0,5644 0,5128 GeneID ab initio 0,4852 0,4632 0,4742 SNAP ab initio 0,5640 0,4297 0,4969 Augustus con suggerimenti RNA-seq 0,5656 0,6727 0,6192 GeneID con suggerimenti RNA-seq 0,4884 0,4639 0,4762 Confronto degli allineamenti e delle predizioni contro il dataset di riferimento (r. esoniche) Evidenze Sensibilità Specificità Accuratezza EST 0,9342 0,6054 0,7698 Proteine omologhe 0,1732 0,9203 0,5468 RNA-seq 0,7334 0,6413 0,6874 Augustus ab initio 0,4489 0,8022 0,6256 GeneID ab initio 0,5245 0,7744 0,6495 SNAP ab initio 0,5459 0,6688 0,6074 Augustus con suggerimenti RNA-seq 0,5078 0,8502 0,6790 GeneID con suggerimenti RNA-seq 0,5296 0,7413 0,6355 Confronto degli allineamenti e delle predizioni contro il dataset di riferimento (giunzioni) Evidenze Sensibilità Specificità Accuratezza EST 0,5566 0,4747 0,5157 Proteine omologhe 0,2493 0,8794 0,5644 RNA-seq 0,8723 0,9507 0,9115 Augustus ab initio 0,6260 0,8347 0,7304 GeneID ab initio 0,6881 0,7536 0,7209 SNAP ab initio 0,5840 0,4538 0,5189 Augustus con suggerimenti RNA-seq 0,7875 0,9112 0,8494 GeneID con suggerimenti RNA-seq 0,6943 0,7521 0,7232 Annotazione finale realizzata con Evidence Modeller, che permette di combinare i risultati delle predizioni e delle evidenze sperimentali in un’unica annotazione finale mediante l’assegnazione di pesi. Pesi EVM assegnati Annotazione 1 Annotazione 2 Annotazione 3 EST Proteine RNA-seq Augustus ab initio GeneID ab initio SNAP ab initio Augustus con suggerimenti RNA-seq GeneID con suggerimenti RNA-seq 3 5 3 1 1 1 0 0 Statistiche generali Numero di geni Media della lunghezza dei geni N50 della lunghezza dei geni Media dei numero di esoni per gene Livelli d’indagine 3 5 0 0 0 0 2 0 Annotazione 1 Annotazione 2 Annotazione 3 26.814 26.243 26.211 1.119,90 1.145,90 1.130,56 1.452 1.446 1.434 4,34 4,77 4,72 Annotazione 1 Sensibilità 3 5 0 0 1 1 2 0 Annotazione 2 Specificità Accuratezza Sensibilità Annotazione 3 Specificità Accuratezza Sensibilità Specificità Accuratezza Identificare i loci genici 0,4396 0,6276 0,5336 0,5620 0,6768 0,6194 0,5600 0,6760 0,6180 Identificare le regione esoniche 0,4119 0,8110 0,6115 0,5012 0,8492 0,6752 0,5008 0,8544 0,6776 Identificare le giunzioni esone-introne 0,5698 0,8383 0,7041 0,7768 0,9093 0,8431 0,7769 0,9132 0,8451 Le statistiche generali non sono sufficienti a valutare le differenze tra le diverse predizioni, è necessario valutarne l’accuratezza. È importate definire metriche adeguate per valutare l’accuratezza di una predizione sotto diversi aspetti. Predittori con accuratezza simile per alcuni aspetti, mostrano un grado di accuratezza completamente diverso per altri. Utilizzare RNA-Seq, che sono ottenibili a costi ridotti e in tempi brevi, come suggerimento per i predittori può migliorare sostanzialmente la predizione a seconda del software utilizzato. È possibile realizzare un’annotazione finale con poche predizioni accurate, consentendo un significativo risparmio di tempo computazionale. Valutare ogni singolo passaggio del protocollo di annotazione permette di avere un’annotazione finale ottimizzata.
Scarica
![Alimentazione, squilibrio emozionale e s[...]](http://s2.diazilla.com/store/data/000199131_1-4df9f30b101216c63cae2f89e397f485-260x520.png)