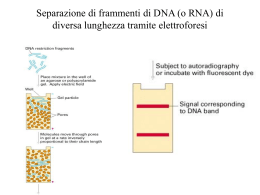
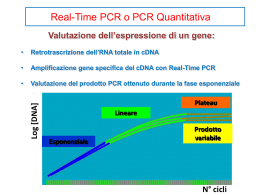
La biologia molecolare nella diagnostica virologica Corso di perfezionamento Nuovi sviluppi nella diagnostica microbiologica e virologica Diagnostica virologica metodi diretti definizione Dimostrazione del virus o suoi componenti Tipo di metodo Ricerca dei virioni al M.E. Ricerca dell’infettività virale Ricerca degli Ag virali Ricerca del genoma virale Ricerca delle sequenze nucleotidiche Quali virus ? difficilmente o non coltivabili (HBV, HCV, papillomavirus, parvovirus B19) a lento sviluppo (CMV, BKV, JCV) pericolosi da coltivare (HIV) emergenti (TTV) Ricerca delle sequenze nucleotidiche Quando ? campioni con la carica virale molto bassa (liquor – HIV, HSV, JCV) distinzione tra infezione latente e attiva (CMV, HSV) riduzione “periodo finestra” (HIV, HCV) monitoraggio delle infezioni croniche valutazione prognostica monitoraggi terapeutici genotipizzazione Ricerca delle sequenze nucleotidiche Ibridazione 100 000 - 10 000 sequenze bersaglio amplificazione ibridazione 10 - 1 sequenze bersaglio Ibridazione degli acidi nucleici reazioni bimolecolari fra una molecola bersaglio (target) e una molecola sonda (probe), in cui catene complementari di acidi nucleici si associano a formare molecole a doppia catena Sonde (probes) sequenze di DNA o di RNA complementari alla sequenza bersaglio che si vuole cercare •acidi nucleici purificati e clonati in vettori genetici •oligonucleotidi di sintesi • sonde generiche o specifiche Sonde “calde” dotate di un sistema di rivelazione 32P, 35S visualizzazione dell’ibrido tramite autoradiografia Sonde “fredde” Fluorochromi fluorescenza Biotina sistemi immunoenziamtici o enzimatici Digossigenina rivelazione colorimetrica o chemiluminescente sistema di rivelazione detection in gene probe assays: (a) enzyme-labeled gene probe (b) hapten-labeled gene probe detected with labeled anti-hapten antibody (c) unlabeled gene probe and hybrid detection by means of anti-hybrid antibody and labeled anti-species antibody Sistema enzimatico Sistema immunoenzimatico Ibridazione in soluzione con successiva cattura dell’ibrido su matrice solida membrana (dot-blot, gel-blot), micropiastra in situ all’interno di cellule, con mantenimento della morfologia cellulare e tessutale Hybrid Capture II ibridazione in fase liquida, cattura su pozzetto rivelazione chemiluminescente Denaturazione Ibridazione Cattura Rivelazione Acquisizione HPV PROBE A: pool di sonde a RNA per HPV-LR: 6, 11, 42, 43 e 44 HPV PROBE B: pool di sonde a RNA per HPV-HR: 16, 18, 31, 33, 35, 39, 45, 51, 52, 56, 58, 59 e 68 Sensiblità 1.000 copie Ibridazione sul filtro (dot-blot) 32P autoradiografia dig coniugatoDigAP X-fosfato + NTB Rivelazione multicolore Metodi di amplificazione metodo substrato thermal cycling PCR target DNA si RT-PCR target RNA si NASBA target RNA no LCR sonda si Q sonda no bPCR segnale no amplificazione del DNA target Polymerase Chain Reaction (PCR) amplificazione di una sequenza di DNA compresa tra due brevi sequenze definite Master mix - 4 dNTP - DNA polimerasi termostabile - 2 oligonucleotidi (primers) •denaturazione •appaiamento •estensione 20-40 volte Polymerase Chain Reaction (PCR) Rivelazione dei prodotti amplificati • presenza della banda • dimensioni della banda • Conferma della specificità - sequenziamento - Southern blotting - analisi con enzimi di restrizione Rivelazione e identificazione di virus polio mediante RT-PCR e tipizzazione mediante RFLP. HaeIII 5 6 7 8 3 DdeI 1 2 3 4 2 DdeI HaeIII DdeI HaeIII 1 M 480 pb 1: Sabin 1 2: Sabin 2 3: Sabin 3 4: “0” RNA 5: virus polio 1 di isolamento clinico 6: virus polio 2 di isolamento clinico 7: virus polio 3 di isolamento clinico 8: 20-100 pb ladder 1: Sabin 1 2: Sabin 2 3: Sabin 3 M: 20-100 bp ladder Rivelazione dei prodotti amplificati PCR/ELISA ampliWell Parvo ID (Microgen) sensibilità: 10 UI standard internazionale (WHO): 1 UI 1 genoma Possibilità di quantificare il prodotto Rivelazione dei prodotti amplificati DEIA (Sorin Biomedica) Rivelazione dei prodotti AMPLICOR® Microwell Plate Test Format Rivelazione dei prodotti •sonde marcate con estere di acridinio (AE) •reattivo di selezione idrolizza AE delle sonde libere •La luce emessa da AE protetto rilevata con luminometro Hybridization Protection Assay (Gene-Probe) Polymerase Chain Reaction (PCR) Reazioni più comunemente usate nel laboratorio diagnostico •PCR singola •PCR nested •PCR multiplex •RT-PCR •Q-PCR (real time PCR) PCR nested amplificazione mediante 2 PCR consecutive: maggiore specificità maggiore sensibilità Primer 1.1 1st Round PCR Primer 1.2 Primer 2.2 2nd Round PCR Primer 2.2 Amplification product PCR multiplex Identificazione di bersagli differenti in una sola reazione • virus differenti • sequenze differenti dello stesso virus Duplex PCR per polyomavirus umani BK e JC RT-PCR Trascrittasi inverse •Moloney murine lekemia virus (M-MLV) •Avian myeloblastosis virus (AMV) •Tth DNA polimerasi - attività di trascrittasi inversa - attività di DNA polimerasi amplificazione • RNA virale genomico • mRNA virali (non distingue tra DNA e RNA) RT/PCR per VP1/ mRNA del parvovirus B19 1 100 Unità di mappa B19/9 B19/9B primers B19/9 5’ GTT TTT TGT GAG CTA ACT AAC A 3’ B19/9B 5’ CCA CGA TGC AGC TAC AAC TT 3’ prodotti ottenuti: 131pb, 251pb, 1615pb RT/PCR per VP1/mRNA del parvovirus B19 1 9 2 10 3 4 5 6 7 8 (1) MWM VI Roche; (2) cellule non infettate (controllo negativo); (3) tempo 0; (4) 24 ore ; (5) 48 ore; (6) 6 giorni; (7,10) campioni bianchi; (8,9) diluizioni scalari della sequenza bersaglio clonata. amplificazione del target RNA Nucleic Acid Sequence – Based Amplification (NASBA) prima reazione di amplificazione alternativa alla PCR (1989) TAS = Transcription - based Amplification System 3SR o SSSR = Self Sustained Sequence Replication amplificazione selettiva dell’RNA attraverso cicli ripetuti di retrotrascrizione e di trascrizione Primer antisenso 5’ utilizza 3 enzimi - sequenza promotore per la T7 RNA polimerasi trascrittasi inversa AMV T7 RNA polimerasi Rnasi H Vantaggi (NASBA vs. RT/PCR) •reazione isotermica •sintesi selettiva del RNA: identificazione virus a RNA, identificazione della riattivazione dei virus latenti •può essere utilizzata in versione quantitativa •elevata sensibilià e specificità Nucleic Acid Sequence – Based Amplification NASBA amplificazione della sonda Ligase Chain Reaction (LCR) Amplificazione di prodotti di ligazione complementari alla sequenza bersaglio Master mix - DNA ligasi termostabile - 4 oligonucleotidi - DNA polimerasi (Gap-LCR) •Denaturazione •Appaiamento, ligazione 25 volte Ligase Chain Reaction (LCR) p1 p2 p3 p4 Ligase Chain Reaction (LCR) Gap – LCR amplificazione della sonda Amplificazione con la Q Replicasi Sonda sequenza complementare alla sequenza bersaglio + Q RNA (MDV-1 RNA) Enzimi • replicasi Q = RNA polimerasi RNA dipendente in grado di duplicare la molecola Q • RNAsi III amplificazione della sonda Amplificazione con la Q Replicasi ibridazione sandwich con una serie di sonde - sonde di cattura (capture probes) - sonde “leganti” (extender probes) - sonde di amplificazione ramificate (bDNA probes) - sonde marcate con enzima (reporter probes) amplificazione del segnale Branched chain DNA (bDNA) •Non richiede la purificazione del campione •Non soggetta alle contaminazioni •Segnale generato dal prodotto della reazione enzimatica è direttamente proporzionale alla carica virale applicazioni quantitative Tecniche molecolari Elevata sensibilità e specificità vantaggi Ricerca di virus difficilmente o non coltivabili Identificazione e tipizzazione Quantificazione del prodotto Facile automazione Rapidità di esecuzione Rischio di contaminazioni falsi positivi limiti Rischio di cross-reazioni con acidi nucleici non specifici Rischio di falsi negativi per inibitori Alti costi
Scarica