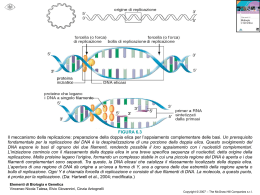
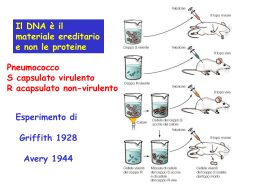
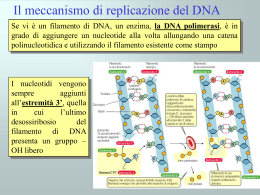
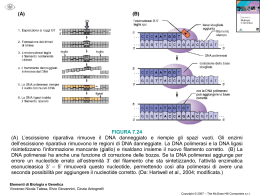
Cap. 3 La replicazione del DNA pp. 66-89 Sintesi 03 • La replicazione del DNA è semiconservativa: un’elica serve da stampo e viene mantenuta invariata, l’altra viene sintetizzata ex novo. • La replicazione richiede l’intervento di enzimi: RNA e DNA polimerasi, ligasi, elicasi e topoisomerasi • Nei procarioti la replicazione procede da un unico punto iniziale, negli eucarioti ciascun cromosoma ha varie origini di replicazione da cui iniziano sintesi simultanee Prova citologica della replicazione semiconservativa Colture di cellule di porcellino d’India Aggiunta di 5-BUdR, analogo della timina, con minore tendenza a legarsi al colorante fluorescente Due cicli di replicazione: 1.TT, TT 2. TU, UT 3.TU, UU Cromosomi arlecchino La replicazione del DNA è semiconservativa Le DNA Polimerasi • Possono aggiungere un nucleotide all’estremità 3’ libera di una catena di acido nucleico, se dispongono di nucleotidi trifosfati • Possono rimuovere un nucleotide all’estremità 3’ o 5’ di una catena di acido nucleico • Non possono iniziare la replicazione se non dispongono di una catena con l’estremità 3’ libera • Non possono legare fra loro due frammenti di DNA Le DNA P di Escherichia coli Nella replicazione di Escherichia coli DNA stampo (elica singola) + primer + dATP, dCTP, dGTP, dTTP una DNA polimerasi DNA a doppia elica 5’---(n-nucl)---3’ + dXTP Energia di legame 5’---(n+1-nucl)---3’ + energia La DNAP III: 1. Allunga la doppia elica in direzione 5’—3’ 2. Controlla l’appaiamento fra basi, e se è imperfetto rimuove l’ultimo nucleotide aggiunto (attività proof-reading) La DNAP I: 1. Degrada l’RNA nella doppia elica in direzione 5’—3’ 2. Allunga la doppia elica, estendendo il frammento in direzione 5’—3’ e rimpiazzando l’RNA stampo La DNA PII ripara il DNA danneggiato Inizio della replicazione; elica leading e elica lagging Primosoma: il complesso costituito da elicasi e RNA polimerasi La “bolla replicativa” La replicazione procede nelle due direzioni a partire dalla forcella Frammenti di Okazaki Meccanismo d’azione delle DNA P III batteriche Alla fine per legare fra loro i frammenti ci vuole la ligasi La ligasi catalizza la formazione di un legame fosfodiestereo Schema dei passaggi enzimatici nella replicazione di E. coli (1) Schema dei passaggi enzimatici nella replicazione di E. coli (2) Schema di replicazione di un cromosoma procariote (nella foto, SV40) Durante la replicazione i superavvolgimenti sono un problema: girasi Durante la replicazione i superavvolgimenti sono un problema: topoisomerasi Schema dei passaggi enzimatici nella replicazione di E. coli 1. 2. 3. 4. 5. 6. 7. 8. La topoisomerasi fa rilassare il filamento Due elicasi (una per ciascuna forca replicativa) denaturano e svolgono un tratto della doppia elica Le single-strand binding proteins stabilizzano il DNA ad elica singola, senza coprire le basi La RNA polimerasi (primasi) si lega all’elicasi e sintetizza un innesco di circa 30 paia di basi La DNA P III lo estende da 5’ a 3’ Ad ogni passaggio la DNA P III rimuove gli appaiamenti sbagliati (proof-reading) La DNA P I degrada l’innesco ed estende il frammento adiacente, procedendo da 5’ a 3’ La ligasi salda i filamenti adiacenti, senza aggiungere alcun nucleotide Due eccezioni ΦX174: DNA a singolo filamento DNA P DNA P Due eccezioni Mosaico del tabacco: RNA DNA P* DNA P * DNA polimerasi RNA dipendente! (trascrittasi inversa) Ciclo cellulare in eucarioti e procarioti (ore) Organismo E. coli Lievito Piante Uomo M 20’ 1 1 G1 25’ 8 8 S 40’ 12 10 Velocità di replicazione: E. coli: 50000 basi al minuto Drosophila: 2600 basi al minuto Topo: 2200 basi al minuto G2 35’ 8 5 Totale 1 2 29 24 I cromosomi eucarioti sono lineari, non circolari Nei cromosomi degli eucarioti ci sono varie unità di replicazione Progressione delle bolle di replicazione lungo il cromosoma eucariote ARS: Autonomous-Replication Sequences Negli eucarioti superiori DNA P α: sintesi dell’elica lagging e del primer DNA P β: riparazione del DNA nucleare DNA P γ: replicazione, solo nei mitocondri DNA P δ: sintesi dell’elica leading DNA P ε: riparazione del DNA nucleare SPECIE Dim. Genoma N repliconi velocità replicaz. E. coli: 4,2 Mb 1 50000 Drosophila: 140 Mb 3500 2600 Topo: 3200 Mb 25000 2200 E alla fine? Telomerasi Le regioni terminali dei cromosomi (telomeri) contengono sequenze ripetute: Ciliati (Tetrahymena): n(TTGGGG) Flagellati (Trypanosoma), uomo: n(TTAGGG) L’enzima telomerasi contiene un tratto di RNA complementare alla sequenza ripetuta e lo usa come primer per replicare l’estremità telomerica 5’ Azione della telomerasi in Tetrahymena L’efficienza della telomerasi è incompleta. La perdita di regioni terminali provoca forme di morte cellulare associate all’invecchiamento Negli eucarioti, sintesi e degradazione di cicline, prodotte da geni-orologio regolano il ciclo cellulare Riassunto 03 • La replicazione del DNA è semiconservativa: un’elica serve da stampo e viene mantenuta invariata, l’altra viene sintetizzata ex novo. • La replicazione richiede l’intervento di enzimi: RNA e DNA polimerasi, ligasi, elicasi e topoisomerasi • Nei procarioti la replicazione procede da un unico punto iniziale, negli eucarioti ciascun cromosoma ha varie origini di replicazione da cui iniziano sintesi simultanee
Scarica