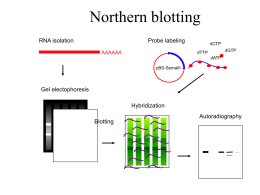
Definizione di genoteca (o library) di DNA Collezione completa di frammenti di DNA, inseriti singolarmente in un vettore di clonaggio. Possono essere di DNA genomico o di cDNA. Materiale di partenza: libraries genomiche Materiale di partenza: cDNA libraries Oligo-dT priming RT First DNA strand 3’ TTTTTTTT 5’ 5’ AAAAAAAA 3’ mRNA Random priming First DNA strand RT RT 5’ 5’ AAAAAAAA 3’ 5’ mRNA Materiale di partenza: cDNA libraries Sintesi del secondo filamento 3’ 5’ First DNA strand TTTTTTTT 5’ AAAAAAAA 3’ mRNA RNAsi H + DNA polimerasi I TTTTTTTT 5’ AAAAAAAA 3’ 3’ 5’ 5’ 5’ pH alcalino TTTTTTTT 5’ AAAAAAAA 3’ 3’ 5’ 3’ 5’ 3’ 5’ 3’ 5’ DNA polimerasi + ligasi First DNA strand TTTTTTTT 5’ AAAAAAAA 3’ Second DNA strand Materiale di partenza: cDNA libraries Sintesi del secondo filamento 3’ First DNA strand 5’ TTTTTTTT 5’ AAAAAAAA 3’ mRNA RNAsi + DNA polimerasi Loop Second DNA strand AAAAAAAA 3’ TTTTTTTT 5’ Nucleasi S1 + DNA polimerasi Second DNA strand 5’ 3’ AAAAAAAA 3’ TTTTTTTT 5’ First DNA strand La sintesi del cDNA produce, a partire da molecole di messaggero uguali, molecole di DNA di diversa lunghezza. Sovrarappresentazione del 3’ se la sintesi è innescata da oligo-dT cDNA-1 (full-lenght) 3’ 5’ TTTTTTTT 5’ AAAAAAAA 3’ mRNA cDNA-2 3’ 5’ 3’ cDNA-3 5’ 3’ 5’ cDNA-4 TTTTTTTT 5’ AAAAAAAA 3’ TTTTTTTT 5’ AAAAAAAA 3’ TTTTTTTT 5’ AAAAAAAA 3’ Vettori plasmidici Vettori fagici Vettori fagici Left Ligasi Concatameri Right Vettori fagici Vettori fagici Il cDNA può essere clonato direzionalmente EcoRI TTTTTTTTGAATTC 5’ AAAAAAAA 3’ 3’ 5’ mRNA dCTP + transferasi terminale TTTTTTTTGAATTC 5’ AAAAAAAA 3’ 3’CCCCCC 5’ RNAsi o pH alcalino TTTTTTTTGAATTC 5’ 3’CCCCCC 5’AAGCTTGGGGGG 3’ Hind III 3’TTCGAACCCCCC 5’AAGCTTGGGGGG Hind III DNA polimerasi TTTTTTTTGAATTC 5’ AAAAAAAACTTAAG 5’ EcoRI Ospiti Batteri Virus (Batteriofagi) Colonie Placche di lisi Screening di una genoteca, ossia come trovare un ago in un pagliaio Screening di una genoteca, ossia come trovare un ago in un pagliaio Screening di una genoteca, ossia come trovare un ago in un pagliaio Problemi tipici e strategie 1. Identificare tutta la sequenza di un gene (o di un mRNA) a partire da un frammento parziale iniziale Sonda 3 Sonda 1 Sonda 2 Sonda 4 Gene A B C E D F G Questa metodica prende il nome di chromosome walking Problemi tipici e strategie 2. Data la sequenza di un gene di una determinata specie, trovare il gene omologo in specie differenti (zoo-blot). 3. Data la sequenza di un gene di una determinata specie, trovare altri geni della stessa specie caratterizzati da sequenze simili. In entrambe i casi l’ibridizzazione va fatta a bassa stringenza. Problemi tipici e strategie 4. Data la sequenza di un peptide di una certa proteina, trovare il cDNA corrispondente. La sonda in questo caso è un oligonucleotide degenerato M I N F P Y G T T T T T C T C 5’ ATGATCAA TT CC TA GG 3’ C C A C A A G G Concetto di rappresentatività Affinchè sia utile, una library deve essere completa, ossia rappresentativa di tutti gli elementi di partenza, siano essi sequenze genomiche o molecole di mRNA. Per quelle genomiche il numero di cloni da cui devono essere composte per avere la rappresentatività voluta è espresso dalla seguente relazione: ln(1 -P) N= ln(1 - f) P è la probabilità voluta f è la frazione del genoma contenuta mediamente in un clone N è il numero di cloni costituenti la library Concetto di rappresentatività Ad esempio, per raggiungere una probabilità del 99% di avere una determinata sequenza in una library genomica umana (3x109 bp) composta da inserti di dimensione media 20000 bp bisogna avere il seguente numero di cloni: ln(1 – 0,99) N= = 690000 ln [1 – (2x104 / 3x109)] Problema: come faccio a identificare tutta la sequenza di un determinato RNA messaggero? mRNA AAAAAAAA 3’ 5’ Cloni di cDNA presenti nella library Sonda In questo caso per identificare la sequenza completa sarebbe sufficiente la strategia del ‘chromosome walking’ Problema: come faccio a identificare tutta la sequenza di un determinato RNA messaggero? mRNA AAAAAAAA 3’ 5’ Cloni di cDNA presenti nella library Sonda In questo caso per identificare la sequenza completa il ‘chromosome walking’ non sarebbe sufficiente. RACE mRNA ? Sequenza nota ? 5’ AAAAAAAA 3’ cDNA 5’ 3’ 3’ 5’ 5’ X * 3’ 5’ 5’ 3’ * Adattatori X 5’ Ligasi * 5’ X * *= 3’ bloccato X 5’ 5’ RACE 5’ mRNA * 5’ 3’ mRNA X * X 3’ 3’ X 5’ 5’ GSP * X X 3’ 5’ X X * 5’ * 5’ 5’ X GSP Amplificazione esponenziale Clonaggio e sequenziamento 5’ 3’ RACE 5’ mRNA * 5’ 3’ mRNA X * 5’ * 5’ X X GSP 5’ * 3’ X 3’ 5’ X Amplificazione esponenziale Clonaggio e sequenziamento 5’ Esempio di clonaggio di un gene Clonaggio del gene a seguito di isolamento della proteina: Identificazione del gene dell’emofilia A (fattore VIII) Gene: 150000 basi mRNA: 7000 basi Proteina: 2351 a.a. Purificazione del fattore VIII di maiale con tecniche biochimiche tradizionali Microsequenza AGGFLMKMFGGHTSREDFCH AHHTFIAAVEQLWDYGMATT FGKRMLKFSSCHTRDEGGAK Sequenze possibili N C C CAU UAU His Tyr Phe CAC UAU 5’ C UUU UUC A C A G C AUU GCU Ile A C G GCU Ala Ala AUC GCA GCG A C G A GUU GAG A CAG A C C G UUU C UGG GAU C UAU A C G GGU C AUG Val Glu Gln Leu Trp Asp Tyr Gly Met GUC GAA CAG CUG UGG GAU mRNA-maiale Sequenza effettiva UAC GGU AUG 3’ Ibridazione di cDNA library di fegato di maiale con un oligonucleotide degenerato 5’ C C CAU UAU His C UUU Tyr Phe A C A G C AUU GCU Ile TGGGATTATGGTATG TGGGACTATGGTATG TGGGATTACGGTATG TGGGACTACGGTATG A C G GCU Ala Ala A C G A GUU GAG A CAG A C C G UUU C UGG GAU C UAU A C G GGU 3’ AUG Val Glu Gln Leu Trp Asp Tyr Gly Met TGGGATTATGGGATG TGGGACTATGGGATG TGGGATTACGGGATG TGGGACTACGGGATG TGGGATTATGGCATG TGGGACTATGGCATG TGGGATTACGGCATG TGGGACTACGGCATG TGGGATTATGGAATG TGGGACTATGGAATG TGGGATTACGGAATG TGGGACTACGGAATG --------CACTATTTCATCGCAGCGGTCGAACAGCTGTGGGATTACGGTATG------------------GTGATAAAGTAGCGTCGCCAGCTTGTCGACACCCTAATGCCATAC------------- Ibridazione di cDNA library di fegato di maiale con un oligonucleotide degenerato TGGGATTATGGTATG TGGGACTATGGTATG TGGGATTACGGTATG TGGGACTACGGTATG TGGGATTATGGGATG TGGGACTATGGGATG TGGGATTACGGGATG TGGGACTACGGGATG TGGGATTATGGCATG TGGGACTATGGCATG TGGGATTACGGCATG TGGGACTACGGCATG TGGGATTATGGAATG TGGGACTATGGAATG TGGGATTACGGAATG TGGGACTACGGAATG --------CACTATTTCATCGCAGCGGTCGAACAGCTGTGGGATTACGGTATG----------- --------GTGATAAAGTAGCGTCGCCAGCTTGTCGACACCCTAATGCCATAC------------- Ibridazione di cDNA library di fegato di maiale con un oligonucleotide degenerato TGGGATTATGGTATG TGGGACTATGGTATG TGGGACTACGGTATG TGGGATTATGGGATG TGGGACTATGGGATG TGGGATTACGGGATG TGGGACTACGGGATG TGGGATTATGGCATG TGGGACTATGGCATG TGGGATTACGGCATG TGGGACTACGGCATG TGGGATTATGGAATG TGGGACTATGGAATG TGGGATTACGGAATG TGGGACTACGGAATG --------CACTATTTCATCGCAGCGGTCGAACAGCTGTGGGATTACGGTATG----------- TGGGATTACGGTATG --------GTGATAAAGTAGCGTCGCCAGCTTGTCGACACCCTAATGCCATAC------------- Clone di cDNA di maiale gagtgcagccaacccgccctgatgaagatgaagcacgtgagcagctttgtccagaagtat E C S Q P A L M K M K H V S S F V Q K Y tccgacaccatagccgagttgcgggagctgcagccgtcggcgagagacttcgaagttcga S D T I A E L R E L Q P S A R D F E V R agccttgtgggctgtggtcacttcgctgaacactatttcatcgcagcggtcgaacagctg S L V G C G H F A E H Y F I A A V E Q L tgggattacggtatgaaaatcatgaagaagaaggctttgctggcccaggaacaggtttca W D Y G M K I M K K K A L L A Q E Q V S tttttcgaggaggagaggaacatattatctcggagcacgagtccttggatcccccagtta F F E E E R N I L S R S T S P W I P Q L Per trovare il gene umano, library genomica o di cDNA? Sonda Human: 269 atgacacctcaacccagtggtccaaaaacatgaaacatttgaccccgagcaccctcacac 328 ||||||| |||||||||||||||||||||||||| Pig: ||||| ||| | | |||||||||| 4022 atgacacttcaacccagtggtccaaaaacatgaactatttggcccagggaaccctcacac 4081 Human: 329 agatagactacaatgagaaggagaaaggggccattactcagtctcccttatcagattg 386 ||||||| || |||||||| || ||| |||||||||||||||| ||| |||||||||| Pig: 4082 agatagagtataatgagaaagaaaaaagggccattactcagtcccccctatcagattg 4139 Screening di una cDNA library umana con la sonda di maiale (ibridizzazione a bassa stringenza) mRNA a b c f d e cDNA consensus umano Screening di library genomiche umane con sonde di cDNA umano mRNA f c Library genomica fago l Library genomica BAC Sequenziamento genomico Perché sequenziare genomi interi ? • Sequenza completa di tutti i geni • Possibilità di determinare la struttura esoni-introni • Mappare i geni e le altre sequenze • Rivelare le regioni di controllo non codificanti • Identificare polimorfismi • Scoprire l’inatteso Approcci sistematici alla identificazione dei geni Sequenziamento delle sole regioni esoniche (Expressed Sequence Tags) cDNA-library plasmidica direzionale Picking singoli cloni 5’ Primer Preparazione DNA 3’ Sequenziamento automatico Deposito in banca dati (300-700 bp) Sequenze EST mRNA (10 copie di cDNA nella library) Inserti AAAAAAAA EST Tratto non sequenziato del clone Craig Venter Francis Collins Sequenziamento genomico Schema approccio tradizionale DNA Genomico Subclonaggio in vettori BAC, P1 o PAC e assemblaggio di contigs con minimo di ridondanza Subclonaggio in vettori da sequenziamento Sequenziamento Sequenziamento genomico Schema whole genome shotgun DNA Genomico Frammenti casuali lunghi (5-20 kb) e corti (0.4-1.2 kb) derivanti da rottura meccanica del DNA clonati in vettori da sequenziamento Sequenziamento automatico bidirezionale Ricostruzione computerizzata della sequenza genomica Contigs: tratti di sequenza assemblati senza discontinuità. Scaffolds: serie di due o più contigs uniti da lunghi inserti le cui estremità sono in diversi contig ma di cui non si conosce la regione centrale. Contig Contig Scaffold Sequenziamento del genoma di Hemophilus Influenzae (1.8 Mb) • 20000 frammenti di 1,6-2 kb • 30000 saggi di sequenziamento • 11,6 Mb di sequenza totale • 30 ore per assemblare la sequenza su un calcolatore con 512 Mb di RAM Per dimostrare che era possibile sequenziare un genoma complesso con il metodo ‘shotgun’, e che avrebbe potuto sequenziare il genoma umano in 2 anni, nel 1998 Venter ha sequenziato il genoma di Drosophila in soli 6 mesi, in collaborazione con il consorzio pubblico di Drosophila Inizialmente si stimava che la sequenza sarebbe stata completa nel 2005, poi nel 2003. La combinazione dell’approccio del consorzio pubblico con quello di Venter hanno portato alla pubblicazione di due sequenze (più o meno indipendenti) nel 2000 E dopo il sequenziamento? >tgcagccaacccgccctgatgaagatgaagcacgtgagcagctttgtccagaagtattccgaca ccatagccgagttgcgggagctgcagccgtcggcgagagacttcgaagttcgaagccttgtgggc tgtggtcacttcgctgaacactatttcatcgcagcggtcgaacagctgtgggattacggtatgaa aatcatgaagaagaaggctttgctggcccaggaacaggtttcatttttcgaggaggagaggaaca tattatctcggagcacgagtccttggatcccccagttatccgacaccatagccgagttgcgggag ctgcagccgtcggcgagagacttcgaagttcgaagccttgtgggctgtggtcacttcgctgaaca ctatttcatcgcagcggtcgaacagctgtgggattacggtatgaaaatcatgaagaagaaggctt tgctggcccaggaacaggtttcatttttcgaggaggagaggaacatattatctcggagcacgagt ccttggatcccccagttagagtgcagccaacccgccctgatgaagatgaagcacgtgagcagctt tgtccagaagtattccgacaccatagccgagttgcgggagctgcagccgtcggcgagagacttcg aagttcgaagccttgtgggctgtggtcacttcgctgaacactatttcatcgcagcggtcgaacag ctgtgggattacggtatgaaaatcatgaagaagaaggctttgctggcccaggaacaggtttcaag ccttgtgggctgtggtcacttcgctgaacactatttcatcgcagcggtcgaacagctgtgggatt acggtatgaaaatcatgaagaagaaggctttgctggcccaggaacaggtttcagagtgcagccaa cccgccctgatgaagatgaagcacgtgagcagctttgtccagaagtattttttcgaggaggagag gaacatattatctcggagcacgagtccttggatcccccagttagagtgcagccaacccgccctga tgaagatgaagcacgtgagcagctttgtccagaagtattccgacaccatagccgagttgcgggag ctgcagccgtcggcgagagacttcgaagttcgaagccttgtgggctgtggtcacttcgctgaaca ctatttcatcgcagcggtcgaacagctgtgggattacggtatgaaaatcatgaagaagaaggctt tgctggcccaggaacaggtttcatccgacaccatagccgagttgcgggagctgcagccgtcggcg agagacttcgaagttcgaagccttgtgggctgtggtcacttcgctgaacactatttcatcgcagc ggtcgaacagctgtttttcgaggaggagaggaacatattatctcggagcacgagtccttggacg> Il problema dell’annotazione del genoma
Scarica