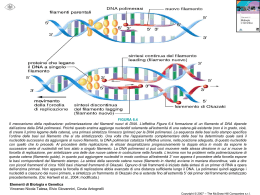
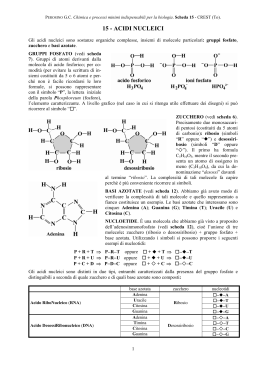
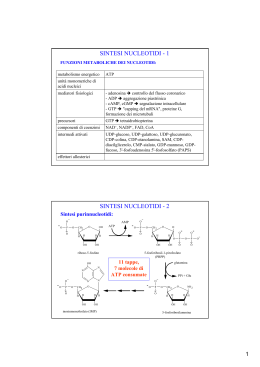
esperto prof. Ciro Formica Immagini e testi tratti dai website di: genome.wellcome.ac.uk, dnaftb.org, unipv.it, unimi.it, wikipedia.it, unibs.it, unisi.it, unina.it, uniroma2.it, nih.gov, zanichelli.it, sciencemag.org, ncbi.gov Nucleotidi Esteri fosforici di un nucleoside formati dalla funzione alcolica –OH di un pentoso (ribosio o deossiribosio) esterificata con una o più molecole di acido fosforico + legame N-glicosidico con una base azotata (purina o pirimidina). Il termine “estere” viene in genere usato per indicare gli esterialcol+acidi organici, ma si estende anche agli esterialcol+ acidi inorganici Esempi Acido+ Base forte Sale Acido organico+ alcol Acido inorganico+ alcol Estere organico Estere inorganico CH3-COOH + NaOH Prodotto finale CH3-COO-Na Acetato di sodio CH3-COOH + CH3CH2-OH CH3-COO-CH2CH3 Acetato di etile Acido fosforico + etanolo Fostato di etile Viene eliminata una molecola di H2O come nelle condensazioni 2 ADENINA In posizione 6: NH2 CITOSINA In posizione 6: NH2 In posizione 4: C=O GUANINA In posizione 2: NH2 In posizione 6: C=O URACILE In posizione 4 e 6: C=O TIMINA In posizione 1: CH3 In posizione 4 e 6: C=O 3 Zuccheri: ribosio e deossiribosio Numerazione degli zuccheri: in senso orario da 1’ a 5’, I primi 4 carboni sull’anello, il 5 fuori dall’anello. Nel DEOSSIRIBOSIO manca un atomo di OSSIGENO in posizione 2’ 4 Il legame tra il ribosio C5’ e il fosfato è un processo di ESTERIFICAZIONE, che avviene tra un gruppo acido (fosfato) e un ossidrile (del ribosio), con eliminazione di una molecola di H2O. Il legame tra ribosio C1’ e base azotata avviene con l’azoto N9. 5 A G C T A/T G/C A+G/ C+T %G+ C Uomo 31,0 19,1 18,4 31,5 0,98 1,04 1,00 37,5 Topo 28,6 21,4 21,7 28,4 1,01 0,99 1,00 43,1 Drosophila 27,3 22,5 22,5 27,6 0,99 1,00 0,99 45,0 Mais 25,6 24,5 24,6 25,3 1,01 1,00 1,00 49,9 E. coli 26,0 24,9 25,2 23,9 1,09 0,99 1,04 50,1 Percentuale di basi azotate nei DNA di varie specie Dati di Franklin Distanza tra 2 eliche Dati di Pauling 2 nm In caso di legame purina-purina > 2 nm In caso di legame pirimidina-pirimidina < 2 nm appaiamento Dati di Chargaff i filamenti formano una spirale , come per le proteine A-T (31%) G-C (19%) Negli acidi nucleici solo radioattività da 32P 6 Da sinistra: Francis Crick e James Watson nel 1953 davanti al modello di doppia elica 7 oltre l’80% di DNA nelle cellule eucariotiche è organizzato sotto forma di nucleosomi ripetuti ognuno dei quali consiste di 200 bp di DNA associate ad un ottametro definito “core” istonico (H2A, H2B, H3, H4) ed un istone “linker” H1. L’ottamero è organizzato in: tetramero H32-H42 posto al centro del NUCLEOSOMA + 2 eterodimeri H2A, H2B attorno al tetramero. Gli istoni hanno p.m. 11-16 kD, H3 e H4 sono conservati dall’evoluzione. Il DNA è organizzato intorno agli istoni con superelica sinistrorsa, 1,65 giri e 10-11 bp. proprietà Filamento guida (leading strand) Filamento ritardo (lagging strand) Aggiunta nucleotidi continua (concorde alla forcella) discontinua (opposta alla forcella) Direzione 5’ 3’ 5’ 3’ Okazaki 100-200 nucleotidi Primer (innesco) sintetizzato da RNA primasi Punto di attacco Telomeri DNA polimerasi 5’-TTAGGG3’ X X X unico numerosi X proofreading Sul filamento ritardo non si completa l’ultimo frammento di Okazaki, perciò il cromosoma tende ad accorciarsi, ma i TELOMERI, brevi sequenze alle estremità dei cromosomi 5’TTAGGG3’, proteggono dall’accorciamento, ma solo per 40-50 duplicazioni cellulari, finché la cellula va in APOPTOSI e muore. Lò’enzima TELOMERASI sintetizza il DNA telomerico perso durante la duplicazione. 9 Attività Azione svolta Elicasi Srotola l'elica e taglia i legami a idrogeno tra le basi complementari. Primasi Sintesi di una piccola catena ribonucleotidica che fa da innesco o primer (lunghezza 9-10 nucleotidi) necessaria per promuovere l’aggiunta di altri nucleotidi. Polimerasi Aggiunta di 1 nucleotide per volta al 3' OH della catena in accrescimento Ligasi Unisce i frammenti neosintetizzati (dopo la rimozione enzimatica degli RNA-primer). Nucleasi Corregge gli eventuali errori nell'aggiunta dei nucleotidi Nelle nostre cellule la frequenza di errori nell'incorporazione delle basi è minima, . dell'ordine di 10-9 -10-10 (un errore ogni miliardo – uno ogni 10 miliardi di basi), 10 Le molecole di DNA in un gradiente di densità ottenuto centrifugando a lungo una soluzione di CsCl galleggiano al punto del gradiente che corrisponde alla loro densità. Per distinguerle, le eliche nuove venivano marcate con 14N (azoto leggero meno denso), le vecchie con 18N (pesante, più denso). Dopo la centrifugazione apparivano dei picchi di densità distinti modalità semiconservativa, cioè che ogni doppia elica formata da un’emielica vecchia e una nuova. filamento lento (laggng) e filamento guida (leading) Conclusioni: dopo 2 replicazioni spariscono le eliche “pesanti” e si trovano solo le leggere la replicazione è semiconservativa. 11 12 tRNA Se il tRNA contiene una Inosina nell’anticodone in wobble il tRNA può leggere codoni nel mRNA che hanno A, U oppure C nella terza posizione. Nella figura è rappresentato l’esempio della Glicina (Gly) 13 Regolazione trascrizione nei procarioti- gli operoni Triptofano. Componenti: operatore (con repressore e co-repressore), promotore, gene regolatore. I geni strutturali dell’operone triptofano (corepressore) codificano per gli enzimi che catalizzano la sua sintesi. Quando il triptofano è presente, non è necessario produrlo: il repressore si lega all’operatore e la trascrizione è bloccata. Il fine è esprimere oppure no il prodotto finale triptofano Lattosio. Componenti: operatore (con induttore), promotore, gene regolatore I geni strutturali dell’operone lattosio (induttore) codificano per gli enzimi per la sua demolizione. Quando è presente il lattosio, il repressore libera l’operatore e la trascrizione è attivata. Il fine è esprimere oppure no il prodotto finale lattosio Splicing: eliminare gli introni lasciando solo gli esoni (porzioni codificanti), poi riassemblare gli esoni. Il fine del differenziamento cellulare è la regolazione genica differenziale, cioè esprimere le proteine (proteoma) che interessano un dato tessuto, organo o 15 apparato La crescita batterica è esponenziale: dopo n generazioni ci saranno 2n batteri. Esempio in 20 generazioni (circa 3 giorni) 220 = 1.048.576 batteri Bacilli 16 DNA stampo 1° ciclo 2° ciclo 3° ciclo 2 copie 4 copie 8 copie 17 18 19
Scarica