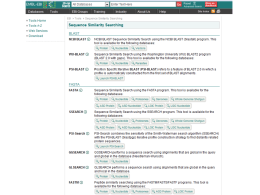
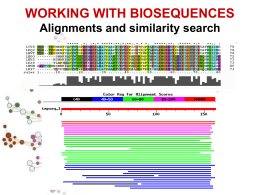
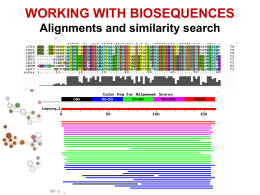
Ricerca di similarità SIMILARITA’ ? OMOLOGIA OMOLOGIA proprieta’ di caratteri (sequenze) dovuta alla loro derivazione dallo stesso antenato comune SIMILARITA’ “grado” di somiglianza tra 2 sequenze •La similarita’ osservata tra due sequenze PUO’ indicare che esse siano omologhe, cioe’ evolutivamente correlate •La similarita’ e’ una proprieta’ quantitativa, si puo’ misurare •L’omologia e’ una proprieta’ qualitativa •Percentuale di omologia Omologia e Analogia, Ortologia e Paralogia OMOLOGIA (ANTENATO COMUNE) ORTOLOGIA PARALOGIA Processo di speciazione Duplicazione genica Descrivo le relazioni tra geni di una famiglia intraorganismo (paralogia) o tra diversi organismi (ortologia) MATRICI PAM (Dayhoff et al. 1978) PAM Point Accepted Mutation Matrici compilate in base all’analisi delle sostituzioni osservate in un dataset costituito da diversi gruppi di sequenze di proteine omologhe 1572 sostituzioni osservate in 71 allineamenti di 71 gruppi di sequenze altamente simili (85%) L’alta conservazione tra le sequenze considerate rendeva non necessaria la correzione per multiple hits (es. A->G->A o A>G->N) L’analisi di questi allineamenti ha mostrato che diverse sostituzioni amminoacidiche vengono osservate con frequenze diverse. MATRICI PAM Le sostituzioni che non influiscono pesantemente sulla funzione della proteina sono “accettate” dalla selezione (Point Accepted Mutation mutazione puntiforme accettata PAM) La frequenza osservata per ciascuna specifica sostituzione (es. A N) puo’ essere usata per stimare la probabilita’ della transizione corrispondente in un allineamento di proteine omologhe. Le probabilita’ di tutte le possibili sostituzioni sono riportate nella matrice PAM La distanza evolutiva di 1 PAM indica la probabilita’ che un residuo muti durante una distanza evolutiva in cui viene accettata una mutazione su 100 residui) MATRICI PAM Dopo aver raccolto le frequenze di mutazione corrispondenti a 1 PAM, le matrici per distanze evolutive superiori possono essere estrapolate dalla matrice PAM1 matrice PAM250 Le matrici PAM sono utili quando si comparano proteine molto simili. E’ necessario che i programmi per la ricerca di similarita’ siano efficaci anche quando si analizzano sequenze non molto conservate. Matrici BLOSUM (Henikoff and Henikoff, 1992) Matrici di sostituzione derivate dall’analisi di oltre 2000 blocchi di allineamenti multipli di sequenze, che riguardavano regioni conservate di sequenze correlate. Per ridurre il contributo di coppie di amminoacidi di proteine altamente correlate, gruppi di sequenze molto simili sono state trattate come se fossero sequenze singole ed e’ stato calcolato il contributo medio di ciascuna posizione. Utilizzando diversi cut-off per il raggruppamento di sequenze simili si sono ottenute diverse matrici BLOSUM. BLOSUM62, BLOSUM80, … L’utilizzo della matrice di similarita’ appropriata per ciascuna analisi e’ cruciale per avere buoni risultati. Infatti relazioni importanti da un punto di vista biologico possono essere indicate da una significativita’ statistica anche molto debole. Allineamento GLOBALE e LOCALE GLOBALE considera la similarita’ tra due sequenze in tutta la loro lunghezza LOCALE considera solo specifiche REGIONI simili tra alcune parti delle sequenze in analisi Algoritmo di Needleman & Wunsch allineamento globale Algoritmo di Smith & Waterman allineamento locale . Ricerca di similarità Tra una sequenza ... e ...un database di sequenze •La similarita’ e’ una proprieta’ statistica, che si evince dalla comparazione di sequenze A COPPIE •Ad ogni comparazione si da’ un punteggio, che aumenta con l’aumentare della similarita’ •Si puo’ discriminare tra matches reali e artefatti stimando la probabilita’ che il match osservato venga ottenuto per caso Ricerca di similarità Tra una sequenza e un database di sequenze •Ovvero, data una sequenza, ci sono cose simili nel database? Ovviamente si cerca di recuperare informazioni ottenute magari per altre specie o altri geni per capire il tratto di DNA che si sta studiando. •Per capire la funzione del gene oppure di fare analisi filogenetiche. Perché trovare somiglianze? Ho trovato un nuovo gene o proteina? Il gene ha somiglianze con qualche altro gene nella stessa specie o in altre specie? Devo trovare le regione di sovrapposizione tra sequenze contigue Voglio studiare l’evoluzione di popolazioni o specie PROGRAMMI PER LA RICERCA DI SIMILARITA’ FastA (Lipman and Pearson, 1985) Il primo algoritmo usato per database similarity searching. Fa uno scanning del database selzionato e cerca brevi “parole” (K-tuples) in comune tra la sequenza in analisi e sequenze dal database (lunghezza 1 o 2 per aa, fino a 5 per nucleotidi). Tutte le parole identificate si trovano sulle diagonali di una immaginaria matrice di comparazione. Le diagonali che presentano la massima densita’ di K-tuples vengono selezionate ed indicano le regioni con la massima similarita’ locale. Negli steps successivi, i punteggi di similarita’ per ciascuna regione vengono ricalcolati dopo l’inserzione di brevi gaps o sostituzioni, attraverso l’utilizzo della matrice di similarita’ appropriata. La diagonale con il punteggio piu’ alto rappresenta la “regione primaria di similarita’”. In seguito, se possibile, eventuali regioni selezionate in precedenza vengono ricongiunte alla regione primaria per formare un unio allineamento. Fase di ottimizzazione. Le sequenze del database vengono ordinate secondo punteggi di similarita’ decrescenti con la sequEnza in analisi. PROGRAMMI PER LA RICERCA DI SIMILARITA’ BLAST Basic Local Alignment Search Tool (Altschul 1990) L’ algoritmo di BLAST e’ euristico, ricerca nel database “parole” di lunghezza almeno “W” con un punteggio di similarita’ di almeno “T” una volta allineate con la sequenza “query” (HSP, High Scoring Pairs). Le “parole” selezionate vengono estese, se possibile, fino a raggiungere un punteggio superiore a “S” oppure un “E-value” inferiore al limite specificato. Come si attribisce il punteggio di similarita’ ad uno specifico allineamento ? Come somma dei punteggi di ciascuna posizione Il punteggio di un match si calcola utilizzando le MATRICI DI SOSTITUZIONE Es. BLOSUM 62: matrice 20x20 in cui ad ogni possibile identita’ o sostituzione viene assegnato un punteggio che si basa sulle frequenze con cui queste si presentano in allineamenti di proteine correlate -Unitary scoring matrix (identity matrix) -PAM Le matrici PAM sono basate su allineamenti globali di sequenze strettamente correlate PAM1 e’ la matrice calcolata con sequenze che presentano l’1% di divergenza. Le altre sono estrapolate da PAM1 -BLOSUM Le matrici BLOSUM sono basate su allineamenti locali. BLOSUM 62 calcolata a partire dall’allineamento di sequenza con almeno il 62% di divergenza Tutte le matrici BLOSUM sono basate su osservazioni, niente estrapolazione -PSSM (position-specific scoring matrices) http://www.blc.arizona.edu/courses/bioinformatics/book_pages/pro-pssms.html Sequenze poco divergenti PAM1 BLOSUM80 molto divergenti BLOSUM62 PAM120 BLOSUM45 PAM250 I gaps hanno punteggio negativo. gap penalty W=a+b(k-1) a = gap opening penalty bk = gap extension fino a lunghezza k La SIGNIFICATIVITA’ di un allineamento si calcola come P value o E value P value e’ la probabilita’ di ottenere un allineamento con punteggio uguale o migliore di quello osservato Si calcola mettendo in relazione il punteggio osservato (S) con la distribuzione attesa di HSP quando si comparano sequenze random della stessa lunghezza e composizione di quella in analisi (query sequence) Piu’ il Pvalue e’ vicino a 0 piu’ e’ significativo 2x10-245 e’ meglio do 0.001 !!! E value e’ il numero atteso di allineamenti con punteggio uguale o migliore di quello osservato Piu’ e’ basso piu’ e’ buono BLAST: Peptide Sequence Databases nr All non-redundant GenBank CDS translations+PDB+SwissProt+PIR+PRF month All new or revised GenBank CDS translation+PDB+SwissProt+PIR+PRF released in the last 30 days. swissprot Last major release of the SWISS-PROT protein sequence database (no updates) Drosophila genome Drosophila genome proteins provided by Celera and Berkeley Drosophila Genome Project (BDGP). yeast Yeast (Saccharomyces cerevisiae) genomic CDS translations ecoli Escherichia coli genomic CDS translations pdb Sequences derived from the 3-dimensional structure from Brookhaven Protein Data Bank kabat [kabatpro] Kabat's database of sequences of immunological interest alu Translations of select Alu repeats from REPBASE, suitable for masking Alu repeats from query sequences. It is available by anonymous FTP from ncbi.nlm.nih.gov (under the /pub/jmc/alu directory). BLAST: Nucleotide Sequence Databases nr All GenBank+EMBL+DDBJ+PDB sequences (but no EST, STS, GSS, or phase 0, 1 or 2 HTGS sequences). No longer "non-redundant". month All new or revised GenBank+EMBL+DDBJ+PDB sequences released in the last 30 days. Drosophila genome Drosophila genome provided by Celera and Berkeley Drosophila Genome Project (BDGP). dbest Database of GenBank+EMBL+DDBJ sequences from EST Divisions dbsts Database of GenBank+EMBL+DDBJ sequences from STS Divisions htgs Unfinished High Throughput Genomic Sequences: phases 0, 1 and 2 (finished, phase 3 HTG sequences are in nr) gss Genome Survey Sequence, includes single-pass genomic data, exon-trapped sequences, and Alu PCR sequences. yeast Yeast (Saccharomyces cerevisiae) genomic nucleotide sequences E. coli Escherichia coli genomic nucleotide sequences pdb Sequences derived from the 3-dimensional structure from Brookhaven Protein Data Bank kabat [kabatnuc] Kabat's database of sequences of immunological interest vector Vector subset of GenBank(R), NCBI, in ftp://ncbi.nlm.nih.gov/blast/db/ mito Database of mitochondrial sequences alu Select Alu repeats from REPBASE, suitable for masking Alu repeats from query sequences. It is available by anonymous FTP from ncbi.nlm.nih.gov (under the /pub/jmc/alu directory). See "Alu alert" by Claverie and Makalowski, Nature vol. 371, page 752 (1994). epd Eukaryotic Promotor Database found on the web at http://www.genome.ad.jp/dbget-bin/www_bfind?epd BLAST: PROGRAMMI blastp compares an amino acid query sequence against a protein sequence database blastn compares a nucleotide query sequence against a nucleotide sequence database blastx compares a nucleotide query sequence translated in all reading frames against a protein sequence database tblastn compares a protein query sequence against a nucleotide sequence database dynamically translated in all reading frames tblastx compares the six-frame translations of a nucleotide query sequence against the six-frame translations of a nucleotide sequence database. Please note that tblastx program cannot be used with the nr database on the BLAST Web page. Nucleotide BLAST Standard nucleotide-nucleotide BLAST blastn Input: sequenza in formato FASTA, GenBank Accession numbers o GI numbers Comparazione con DATABASE NUCLEOTIDICI: dbEST, ... nr, MEGABLAST usa un algoritmo greedy (ingordo) veloce ed ottimizzato per comparare sequenze che differiscono poco Search for short nearly exact matches blastn con parametri scelti in modo da ottimizzare la ricerca di matches quasi esatti e brevi. Questi si trovano spesso per caso, percio’ utilizza alto Evalue, piccola dimensione della parola e filtering. Protein BLAST Standard protein-protein BLAST blastp Input: sequenza in formato FASTA, GenBank Accession numbers o GI numbers Comparazione con DATABASE PROTEICI: nr(translated), swissprot, ... PSI-BLAST - Position Specific Iterated BLAST usa una ricerca iterativa in cui le sequenze trovate con la prima ricerca vengono utilizzate per costrutire un modello per i punteggi di similarita’ da utilizzare per le ricerche successive. I risultati di ciascuna iterazione vengono utilizzati per rifinire il profilo. Questa strategia aumenta la sensibilita’ della ricerca e la sua predittivita’ in termini funzionali. Di solito si usano matrici di sostituzione AxA dove A e’ la dimensione dell’alfabeto. PSI-BLAST usa Una matrice QxA dove Q e’ la lunghezza della query sequence; in ogni posizione, il costo di una lettera dipende dalla posizione stessa e dalla lettera nella query sequence. PHI-BLAST - Pattern Hit Initiated BLAST Translating BLAST Translating BLAST attraverso la traduzione concettuale della query sequence o dei database permette di comparare una seqeunza nucleotidia con database di proteine o viceversa. Translated query - Protein db: blastx la query (seq. Nucleotidica) viene tradotta in tutti i sei possibili frames e la sequenza proteica ottenuta viene comparata con i database di proteine. Protein query - Translated db tblastn protein query sequence comparata con la traduzione dei database nucleotidici Translated query - Translated db tblastx query seq. nucleotidica tradotta comparata con la traduzione dei database nucleotidici Search for conserved domains Search the Conserved Domain Database using RPS-BLAST Search by domain architecture [DART] CD-Search compares a protein sequence against the Conserved Domain Database with the RPS-BLAST program. This database currently contains domains derived from two popular collections, Smart and Pfam, plus contributions from colleagues at NCBI. This allows known functional and structural domains to be identified on protein query sequences. Pairwise BLAST Comparazione tra due sequenze utilizzando l’algoritmo di BLAST blastn - for nucleotide - nucleotide comparisons blastp - for protein - protein comparisons tblastn - compares the protein "Sequence 1" against the nucleotide "Sequence 2" which has been translated in all six reading frames blastx - compares the nucleotide "Sequence 1" against the protein "Sequence 2" tblastx - compares nucleotide "Sequence 1" translated in all six reading frames against the nucleotide "Sequence 2" translated in all six reading frames. Genomic BLAST pages Human Genome Microbial Genomes Arabidopsis thaliana Other eukaryotes Specialized BLAST pages VecScreen - BLAST-based detection of vector contamination IgBLAST - Analysis of immunoglobulin sequences in GenBank OLD Finished and Unfinished Microbial Genomes
Scarica